Процесс самоудвоения днк: процесс самоудвоения молекулы ДНК называется?
Что такое репликация молекул ДНК и как она осуществляется — Клетка — структурная и функциональная единица жизни
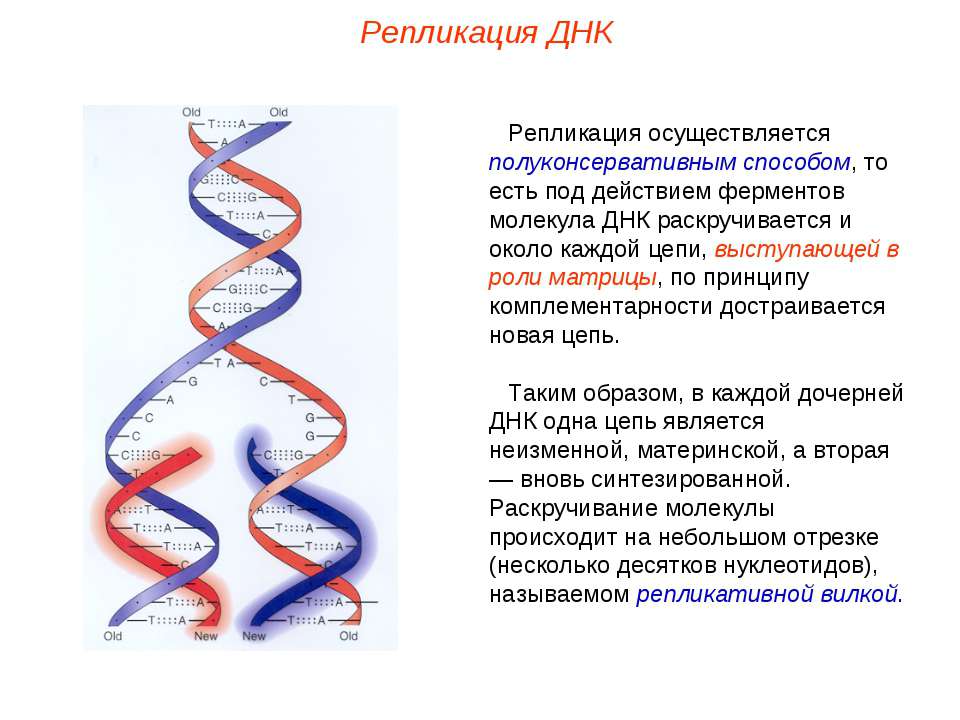
Репликация — это процесс самоудвоения молекул ДНК при участии ферментов. Репликация осуществляется перед каждым клеточным делением. Она начинается с раскручивания спирали ДНК в S-периоде интерфазы под действием фермента ДНК-полимеразы. На каждой из цепей, образовавшихся после разрыва водородных связей, синтезируется по принципу комплементарности и антипараллельности дочерняя цепь ДНК. Причем одна из новых цепей синтезируется сплошной, а вторая — в виде коротких фрагментов, которые затем сшиваются специальным ферментом — ДНК-лигазой.
Таким образом, каждая полинуклеотидная цепь выполняет роль матрицы для новой комплементарной цепи. В каждой из 2-х молекул ДНК одна цепь остается от родительской молекулы, а другая является вновь синтезированной. Такой принцип репликации назван полуконсервативным.
Биологический смысл репликации заключается в точной передаче наследственной информации от материнской клетки к дочерним, что и происходит при делении соматических клеток.
Самая важная особенность репликации ДНК — ее высокая точность.
Если при репликации ДНК последовательность нуклеотидов в ее молекуле нарушается в силу каких-либо причин, то в большинстве случаев эти повреждения устраняются клеткой самостоятельно. Исправление нарушений последовательности нуклеотидов в молекуле ДНК называется репарацией. Изменение происходит обычно в одной из цепей ДНК. Вторая цепь остается неизмененной. Поврежденный участок первой цепи вырезается с помощью ферментов — ДНК-репарирующих нуклеаз. Другой фермент — ДНК-полимераза — копирует информацию с неповрежденной цепи, вставляя необходимые нуклеотиды в поврежденную цепь. Затем ДНК-лигаза сшивает вставленный участок с цепью ДНК. В итоге поврежденная молекула восстанавливается.
Однако бывают случаи, когда пропускается несколько нуклеотидов, или вставляется несколько лишних, или один нуклеотид вставляется вместо другого, например, Ц вместо Т или А вместо Г. Такие изменения последовательности нуклеотидов в молекуле ДНК являются мутациями.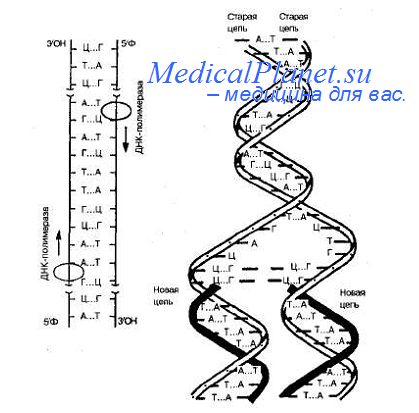 Их воспроизведение в последующих поколениях клеток может приводить к патологии.
Их воспроизведение в последующих поколениях клеток может приводить к патологии.
Самоудвоение молекулы днк
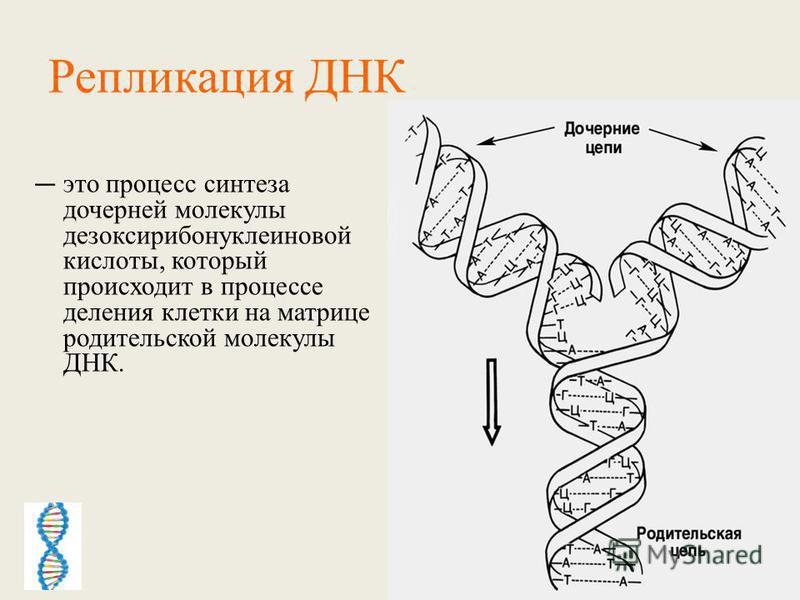
Одним из уникальных свойств молекулы ДНК является ее способность к самоудвоению — воспроизведению точных копий исходной молекулы. Благодаря этой способности молекулы ДНК, осуществляется передача наследственной информации от материнской клетки дочерним во время деления. Процесс самоудвоения молекулы ДНК называют
Репликация
— сложный процесс, идущий с участием
ферментов (ДНК-полимераз) (рис. 276).
Репликация осуществляется полуконсервативным
способом, то есть под действием
ферментов молекула ДНК раскручивается
и около каждой цепи, выступающей в роли
матрицы, по принципу комплементарности
достраивается новая цепь. Таким образом,
в каждой дочерней ДНК одна цепь является
материнской, а вторая — вновь
синтезированной.
В
Рис. 276. Репликация ДНК.
материнской ДНК цепи антипараллельны. ДНК-полимеразы способны двигаться в одном направлении — от 3′-конца к 5′-концу, строя дочернюю цепь антипараллельно — от 5′ к 3′-концу. Поэтому ДНК-полимераза передвигается в направлении 3’→5′ по одной цепи (3′-5′), синтезируя дочернюю. Эта цепь называется Эта цепь называетсяотстающей. Таким образом, на цепи
3′-5′ репликация идет непрерывно, а на
цепи 5′-3′ — прерывисто.
Эта цепь называетсяотстающей. Таким образом, на цепи
3′-5′ репликация идет непрерывно, а на
цепи 5′-3′ — прерывисто.Во время репликации энергия молекул АТФ не расходуется, так как для синтеза дочерних цепей при репликации используются не дезоксирибонуклеотиды (содержат один остаток фосфорной кислоты), а дезоксирибонуклеозидтрифосфаты (содержат три остатка фосфорной кислоты). При включении дезоксирибонуклеозидтрифосфатов в полинуклеотидную цепь два концевых остатка отщепляются, и освободившаяся энергия используется на образование сложноэфирной связи между нуклеотидами.
35.5.5. Рибонуклеиновые кислоты
Молекулы РНК являются полимерами, мономерами которых являются рибонуклеотиды, образованные:
остатком пятиуглеродного сахара — рибозы;
остатком одного из азотистых оснований:
пиримидиновых — урацил, цитозина;
остатком фосфорной кислоты.

Структурная организация рнк
Молекула РНК представляет собой неразветвленный полинуклеотид, имеющий третичную структуру. В отличие от ДНК, она образована не двумя, а одной полинуклеотидной цепочкой. Однако ее нуклеотиды также способны образовывать водородные связи между собой, но это внутри–, а не межцепочечные соединения комплементарных нуклеотидов. Цепи РНК значительно короче цепей ДНК.Информация о структуре молекулы РНК заложена в молекулах ДНК. Синтез молекул РНК происходит на матрице ДНК с участием ферментов РНК-полимераз и называется
Если содержание ДНК в клетке относительно постоянно, то содержание РНК сильно колеблется. Наибольшее количество РНК в клетках наблюдается во время синтеза белка.
Существует три основных класса рибонуклеиновых кислот:
информационная (матричная) РНК — иРНК;
транспортная РНК — тРНК;
рибосомальная РНК — рРНК.

Репликация (самоудвоение) ДНК | Биохимия. Реферат, доклад, сообщение, кратко, презентация, лекция, шпаргалка, конспект, ГДЗ, тест
Перед каждым клеточным делением происходит самоудвоение молекулы ДНК. Процесс удвоения ДНК называется репликацией (лат. replicatio — «повторение»). Он начинается с того, что двойная спираль ДНК начинает раскручиваться, затем в какой-то точке ДНК (или в нескольких точках) обе цепи под действием фермента ДНК-полимеразы расходятся, образуя так называемую репликационную вилку. ДНК в данной части хромосомы удваиваются, образуя две полные дочерние спирали (рис. 61).
Каждая одинарная цепь по принципу комплементарности притягивает к себе свободные нуклеотиды, имеющиеся в клетке, и достраивается до двойной, дополняясь по принципу комплементарности: А-Т и Г-Ц. Таким образом, каждая полинуклеотидная цепь выполняет роль  В итоге вместо одной молекулы ДНК образуются две, точно такие же по нуклеотидному составу, как и первоначальная. При этом каждая из образовавшихся двухцепочечных молекул имеет одну материнскую нить ДНК, а другую — вновь образованную.
В итоге вместо одной молекулы ДНК образуются две, точно такие же по нуклеотидному составу, как и первоначальная. При этом каждая из образовавшихся двухцепочечных молекул имеет одну материнскую нить ДНК, а другую — вновь образованную.
| Рис. 61. Репликация ДНК |
Это лишь схематическое описание процесса самоудвоения ДНК. Материал с сайта http://doklad-referat.ru На самом деле этот процесс очень сложен, в нём участвует множество разных ферментов, способствующих расплетению ДНК, предотвращению её спутывания, а также обеспечивающих сшивание фрагментов ДНК в целостную двойную спираль.
Процесс репликации обеспечивает целая серия белков:
- ДНК-полимераза и РНК-праймаза катализируют полимеризацию нуклеотидов;
- ДНК-геликаза и дестабилизирующие белки расплетают и удерживают спираль;
- ДНК-лигаза и фермент, разрушающий РНК-затравки, сшивают фрагменты;
- ДНК-топоизомераза устраняет суперспираль перед репликативной вилкой;
- инициаторные белки образуют новые репликативные вилки.

Из приведённого неполного перечня участников репликации ДНК можно понять, как осуществляется точность выполнения репликации и какова сложность этого процесса.
На этой странице материал по темам:Репликация днк реферат биохимия скачать
Самоудвоение ДНК. Образование двухроматидных хромосом.(РЕПЛИКАЦИЯ) — Студопедия
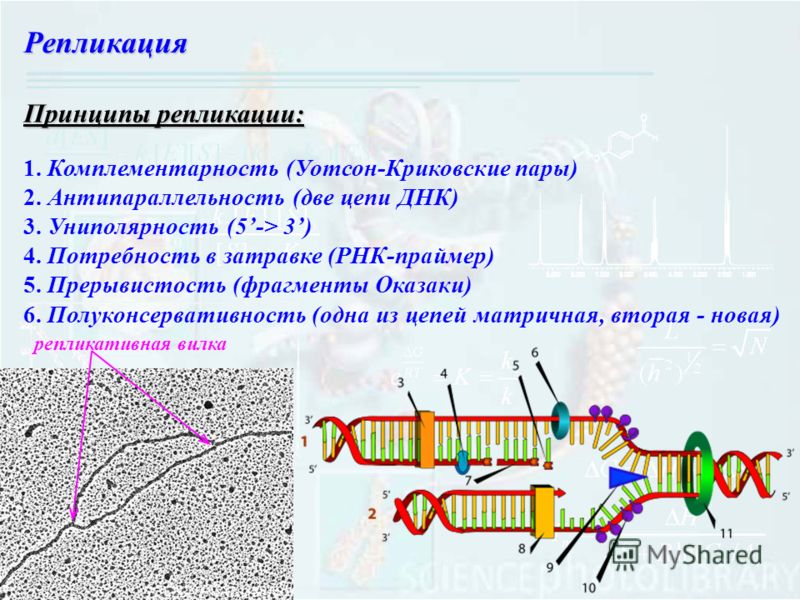
Реплика́ция ДНК — процесс синтеза дочерней молекулы ДНК на матрице родительской молекулы ДНК. В ходе последующего деления материнской клетки каждая дочерняя клетка получает по одной копии молекулы ДНК, которая является идентичной ДНК исходной материнской клетки. Этот процесс обеспечивает точную передачу генетической информации из поколения в поколение. Репликацию ДНК осуществляет сложный ферментный комплекс, состоящий из 15—20 различных белков, называемый
Репликация ДНК — ключевое событие в ходе деления клетки. Принципиально, чтобы к моменту деления ДНК была реплицирована полностью и при этом только один раз. Это обеспечивается определёнными механизмами регуляции репликации ДНК. Репликация проходит в три этапа:
Принципиально, чтобы к моменту деления ДНК была реплицирована полностью и при этом только один раз. Это обеспечивается определёнными механизмами регуляции репликации ДНК. Репликация проходит в три этапа:
1. инициация репликации
2. элонгация
3. терминация репликации.
Регуляция репликации осуществляется в основном на этапе инициации. Это достаточно легко осуществимо, потому что репликация может начинаться не с любого участка ДНК, а со строго определённого, называемого сайтом инициации репликации. В генометаких сайтов может быть как всего один, так и много. С понятием сайта инициации репликации тесно связано понятие репликон. Репликон — это участок ДНК, который содержит сайт инициации репликации и реплицируется после начала синтеза ДНК с этого сайта. Геномы бактерий, как правило, представляют собой один репликон, это значит, что репликация всего генома является следствием всего одного акта инициации репликации. Геномы эукариот (а также их отдельные хромосомы) состоят из большого числа самостоятельных репликонов, это значительно сокращает суммарное время репликации отдельной хромосомы.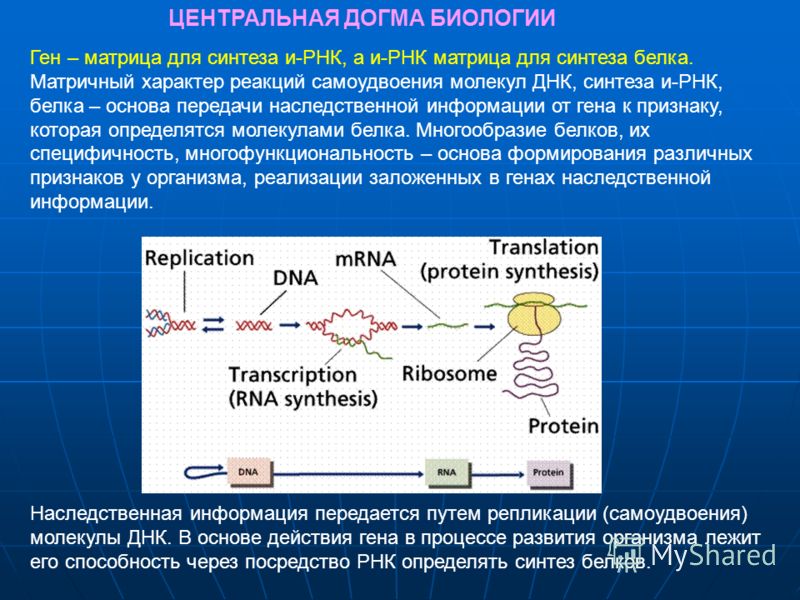 Молекулярные механизмы, которые контролируют количество актов инициации репликации в каждом сайте за один цикл деления клетки, называются контролем копийности. В бактериальных клетках помимо хромосомной ДНК часто содержатся плазмиды, которые представляют собой отдельные репликоны. У плазмид существуют свои механизмы контроля копийности: они могут обеспечивать синтез как всего одной копии плазмиды за клеточный цикл, так и тысяч копий[1].
Молекулярные механизмы, которые контролируют количество актов инициации репликации в каждом сайте за один цикл деления клетки, называются контролем копийности. В бактериальных клетках помимо хромосомной ДНК часто содержатся плазмиды, которые представляют собой отдельные репликоны. У плазмид существуют свои механизмы контроля копийности: они могут обеспечивать синтез как всего одной копии плазмиды за клеточный цикл, так и тысяч копий[1].
Репликация начинается в сайте инициации репликации с расплетания двойной спирали ДНК, при этом формируется репликационная вилка — место непосредственной репликации ДНК. В каждом сайте может формироваться одна или две репликационные вилки в зависимости от того, является ли репликация одно- или двунаправленной. Более распространена двунаправленная репликация. Через некоторое время после начала репликации в электронный микроскоп можно наблюдатьрепликационный глазок — участок хромосомы, где ДНК уже реплицирована, окруженный более протяженными участками нереплицированной ДНК[1].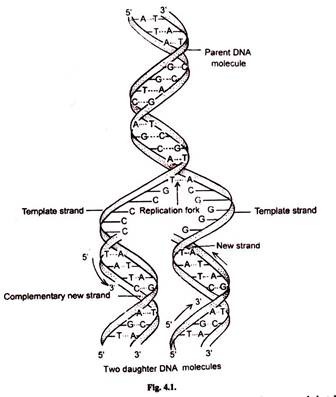
В репликационной вилке ДНК копирует крупный белковый комплекс (реплисома), ключевым ферментом которого является ДНК-полимераза. Репликационная вилка движется со скоростью порядка 100 000 пар нуклеотидов в минуту у прокариот и 500—5000 — у эукарио
Молекулярный механизм репликации
Ферменты (хеликаза, топоизомераза) и ДНК-связывающие белки расплетают ДНК, удерживают матрицу в разведённом состоянии и вращают молекулу ДНК. Правильность репликации обеспечивается точным соответствием комплементарных пар оснований и активностью ДНК-полимеразы, способной распознать и исправить ошибку. Репликация у эукариот осуществляется несколькими разными ДНК-полимеразами. Далее происходит закручивание синтезированных молекул по принципусуперспирализации и дальнейшей компактизации ДНК. Синтез энергозатратный.
Цепи молекулы ДНК расходятся, образуют репликационную вилку, и каждая из них становится матрицей, на которой синтезируется новая комплементарная цепь. В результате образуются две новые двуспиральные молекулы ДНК, идентичные родительской молекуле.
Образование двухроматидных хромасом происходит в анафазе
Механизм репликации смотреть в 37
28 февраля 2003 года исполнилось 50 лет со дня открытия структуры ДНК Френсисом Криком и Джеймсом Уотсоном, которое совершило революцию в биологии и медицине.: Lenta.ru
Начало этой истории можно принять за шутку. «А мы только что открыли секрет жизни!» – сказал один из двоих мужчин, вошедших в кембриджский Игл паб (Eagle pub) ровно 50 лет назад – 28 февраля 1953 года. И эти люди, работавшие в лаборатории неподалеку, нисколько не преувеличивали. Одного из них звали Френсис Крик (Francis Crick), а другого – Джеймс Уотсон (James Watson).
Уотсон и Крик открыли структуру дезоксирибонуклеиновой кислоты (ДНК) – вещества, которое содержит всю наследственную информацию. Через несколько месяцев после исторического заявления в пабе вышла осторожная публикация работы двух исследователей в журнале Nature (Watson J.D., Crick F.H.C. Molecular structure of nucleic acids // Nature. 1953. V. 171. P. 738-740). Статья заканчивалась предположением о том, что открытие структуры ДНК может объяснить механизмы копирования генетического материала.
1953. V. 171. P. 738-740). Статья заканчивалась предположением о том, что открытие структуры ДНК может объяснить механизмы копирования генетического материала.
К пятидесятым годам было известно, что ДНК – большая молекула, которая состоит из тысяч соединенных между собой в линию маленьких молекул четырех разных видов – нуклеотидов. Также ученые знали, что именно ДНК отвечает за хранение и передачу по наследству генетической информации, похожей на текст, написанный алфавитом из четырех букв. Неизвестными оставались пространственная структура этой молекулы и механизмы, по которым ДНК передается по наследству от клетки к клетке и от организма к организму.
В 1948 году Лайнус Полинг (Linus Pauling) открыл пространственную структуру других макромолекул – белков. Прикованный нефритом к постели Полинг несколько часов складывал бумагу, которой он пытался смоделировать конфигурацию белковой молекулы, и создал модель структуры, названной «альфа-спиралью».
По словам Уотсона, после этого открытия в их лаборатории была популярна гипотеза о спиральном строении ДНК.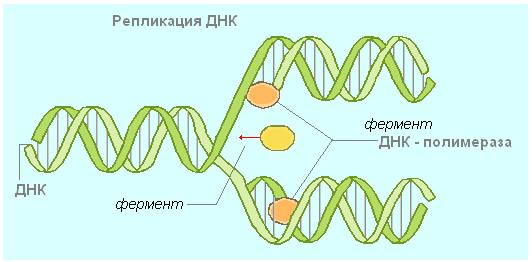 Уотсон и Крик сотрудничали с ведущими специалистами по рентгеноструктурному анализу, а Крик умел практически безошибочно обнаруживать признаки спирали на снимках, полученных таким способом.
Уотсон и Крик сотрудничали с ведущими специалистами по рентгеноструктурному анализу, а Крик умел практически безошибочно обнаруживать признаки спирали на снимках, полученных таким способом.
Полинг тоже считал, что ДНК – спираль, причем, состоящая из трех нитей. Однако, он не мог объяснить ни природы такой структуры, ни механизмы самоудвоения ДНК для передачи дочерним клеткам.
Открытие двуспиральной структуры произошло после того, как Морис Уилкинс (Maurice Wilkins) тайно показал Уотсону и Крику рентгеновский снимок молекулы ДНК, сделанный его сотрудницей Розалинд Франклин (Rosalind Franklin). На этом снимке они четко узнали признаки спирали и направились в лабораторию, чтобы проверить все на объемной модели.
В лаборатории выяснилось, что мастерская не поставила необходимые для стереомодели металлические пластины, и Уотсон вырезал из картона четыре вида макетов нуклеотидов – гуанина (G), цитозина (C), тимина (T) и аденина (A) – и стал раскладывать их на столе. И тут он обнаружил, что аденин соединяется с тимином, а гуанин – с цитозином по принципу «ключ-замок». Именно таким образом соединяются между собой две нити спирали ДНК, то есть напротив тимина из одной нити всегда будет находиться аденин из другой, и ничто иное.
Именно таким образом соединяются между собой две нити спирали ДНК, то есть напротив тимина из одной нити всегда будет находиться аденин из другой, и ничто иное.
Такое расположение позволило объяснить механизмы копирования ДНК: две нити спирали расходятся, и к каждой из них достраивается из нуклеотидов точная копия ее бывшей «партнерши» по спирали. По такому же принципу, как с негатива в фотографии печатают позитив.
Очень печально сложилась судьба Розалинд Франклин. Уилкинс называл свою подчиненную исключительно «синим чулком» и находился с ней в постоянном конфликте. Хоть Франклин и не поддерживала гипотезу о спиральном строении ДНК, именно ее снимки сыграли решающую роль в открытии Уотсона и Крика. И, может, Полинг удостоился бы четвертой Нобелевской премии, если бы он смог увидеть эти снимки раньше, чем британские исследователи.
До премии, которую получили Уилкинс, Уотсон и Крик, Розалинд не дожила. Она скончалась от рака в 1958 году.
Очевидно, что открытие пространственной структуры ДНК совершило революцию в мире науки и повлекло за собой целый ряд новых открытий, без которых нельзя представить не только современную науку, но и современную жизнь в целом
В шестидесятых годах прошлого века предположение Уотсона и Крика о механизме репликации (удвоения) ДНК полностью подтвердилось.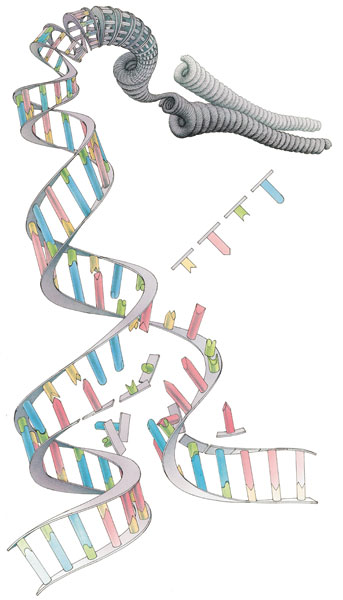 Кроме того, было показано, что в этом процессе принимает участие специальный белок – ДНК-полимераза.
Кроме того, было показано, что в этом процессе принимает участие специальный белок – ДНК-полимераза.
Примерно в то же время было совершено другое важное открытие – генетический код. Как уже говорилось выше, ДНК содержит в себе информацию обо всем, что передается по наследству, в том числе о линейной структуре каждого белка в организме. Белки, как и ДНК, представляют длинные молекулярные цепочки из аминокислот. Этих аминокислот 20. Соответственно, было неясно каким образом «язык» ДНК, состоящий из четырехбуквенного алфавита переводятся на «язык» белков, где используется 20 «букв».
Оказалось, что сочетание из трех нуклеотидов ДНК четко соответствует одной из 20 аминокислот. И, таким образом «написанное» на ДНК однозначно переводится в белок.
В семидесятых годах появились еще два важнейших метода, основанные на открытии Уотсона и Крика. Это секвенирование и получение рекомбинатной ДНК. Секвенирование позволяет «прочитать» последовательность нуклеотидов в ДНК. Именно на этом методе основана вся программа «Геном человека».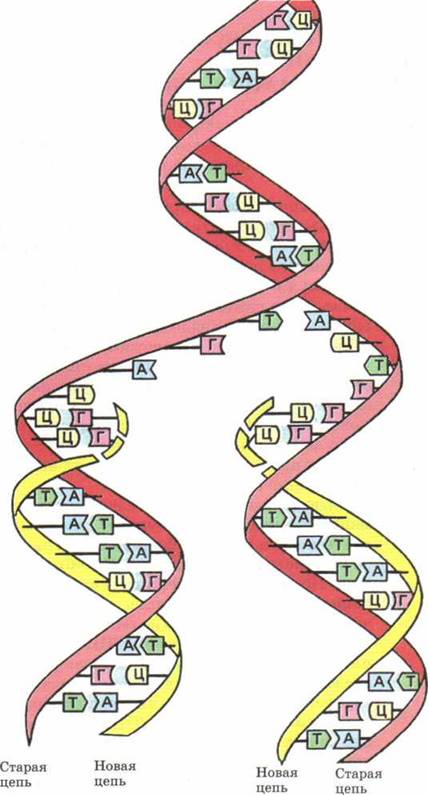
Получение рекомбинантной ДНК по другому называют молекулярным клонированием. Суть этого метода заключается в том, что в молекулу ДНК встраивают фрагмент, содержащий определенный ген. Таким образом, например получают бактерии, которые содержат ген человеческого инсулина. Инсулин, полученный таким способом, называется рекомбинатным. Этим же методом созданы все «генетически модифицированные продукты».
Как ни парадоксально, репродуктивное клонирование, о котором сейчас все говорят, появилось раньше, чем была открыта структура ДНК. Понятно, что сейчас учеными, проводящие такие эксперименты, активно используются результаты открытия Уотсона и Крика. Но, изначально, метод не базировался на нем.
Следующим важным шагом науки стала разработка в восьмидесятых годах полимеразно-цепной реакции. Эта технология используется для быстрого «размножения» нужного фрагмента ДНК и уже нашла множество применений как в науке, так в медицине и технологии. В медицине с помощью ПЦР проводят быструю и точную диагностику вирусных заболеваний. Если в массе ДНК, полученной из анализа пациента, даже в минимальном количестве есть гены, принесенные вирусом, то с помощью ПЦР можно добиться их «размножения» и после этого легко идентифицировать.
Если в массе ДНК, полученной из анализа пациента, даже в минимальном количестве есть гены, принесенные вирусом, то с помощью ПЦР можно добиться их «размножения» и после этого легко идентифицировать.
Кроме того, что открытие Уотсона и Крика стало основой множества научных исследований, включая знаменитый проект «Геном человека», молекула ДНК оставила след в современной живописи, кинематографе, архитектуре.
Пластический обмен. Репликация, ассимиляция, анаболизм
(анаболизм, ассимиляция).
Автор статьи — Л.В. Окольнова.
Проще говоря, это любые процессы и реакции образования, синтеза веществ.
Примеры:
● репликация ДНК
● синтез и-РНК
● синтез белка
● синтез липидов и углеводов
● фотосинтез
● хемосинтез
Подробно каждый из этих процессов мы будем разбирать в соответствующих темах, здесь же рассмотрим исходные вещества, продукты, получающиеся при энергетическом обмене, место синтеза и организмы, в которых происходят эти процессы.
Репликация (самоудвоение) ДНК.
Из одной молекулы получаются 2 и процесс идет с затратами энергии.
Это основной процесс абсолютно для всех живых ( и неживых систем)
● у вирусов (неживых систем) — он происходит в клетках носителя;
● у бактерий — в цитоплазме (прокариоты)
● у всех других эукариотических организмов — в ядре
Синтез и-РНК (транскрипция).
Так же основной, базовый процесс для всего живого.
Базой, матрицей для синтеза молекулы служит ДНК.
У бактерий (прокриотических организмов) — осуществляется в цитоплазме, у всех эукариотов — в ядре.
Синтез белка.
Все живое потребляет и синтезирует белки.
Процесс построения полимера (белка) из мономеров (аминокислот) происходит в рибосомах.
Рибосомы есть абсолютно во всех клетках — как у бактерий, так и у представителей всех царств эукариотического мира.
Процесс многостадийный и требует большого количества энергии:
1. синтез и-РНК на базе ДНК
синтез и-РНК на базе ДНК
2. выход и-РНК из ядра в цитоплазму и прикрепление к рибосоме
3. “считывание” рибосомой информации с и-РНК
4. транспорт соответствующих аминокислот с помощью т-РНК
5. построение белковой нити
Синтез липидов и углеводов.
мономер | полимер |
глицерин и жирные кислоты | липиды |
| моносахариды (глюкоза, фруктоза, рибоза) | полисахариды, т.е. углеводы |
Происходит в эндоплазматической сети.
Фотосинтез.
Это прямо классический пример пластического обмена — из неорганических веществ получаются органические.
● у растений — во всех клетках наземной части организма в хлоропластах
● у бактерий — в пигменте — хлорофилле
Хемосинтез.
Это процесс пластического обмена, характерный исключительно для бактерий.
Репликация ДНК — компоненты репликационной вилки. Реплика
2. Общие представления
Репликация ДНК — ключевое событие в ходе деления клетки. Принципиально, чтобы к моменту деления ДНК была реплицирована полностью и при этом только один раз. Это обеспечивается определёнными механизмами регуляции репликации ДНК. Репликация проходит в три этапа:
- элонгация
- терминация репликации.
- инициация репликации
Регуляция репликации осуществляется в основном на этапе инициации. Это достаточно легко осуществимо, потому что репликация может начинаться не с любого участка ДНК, а со строго определённого, называемого сайтом инициации репликации. В геноме таких сайтов может быть как всего один, так и много. С понятием сайта инициации репликации тесно связано понятие репликон. Репликон — это участок ДНК, который содержит сайт инициации репликации и реплицируется после начала синтеза ДНК с этого сайта. Геномы бактерий, как правило, представляют собой один репликон, это значит, что репликация всего генома является следствием всего одного акта инициации репликации. Геномы эукариот а также их отдельные хромосомы состоят из большого числа самостоятельных репликонов, это значительно сокращает суммарное время репликации отдельной хромосомы. Молекулярные механизмы, которые контролируют количество актов инициации репликации в каждом сайте за один цикл деления клетки, называются контролем копийности. В бактериальных клетках помимо хромосомной ДНК часто содержатся плазмиды, которые представляют собой отдельные репликоны. У плазмид существуют свои механизмы контроля копийности: они могут обеспечивать синтез как всего одной копии плазмиды за клеточный цикл, так и тысяч копий.
Репликон — это участок ДНК, который содержит сайт инициации репликации и реплицируется после начала синтеза ДНК с этого сайта. Геномы бактерий, как правило, представляют собой один репликон, это значит, что репликация всего генома является следствием всего одного акта инициации репликации. Геномы эукариот а также их отдельные хромосомы состоят из большого числа самостоятельных репликонов, это значительно сокращает суммарное время репликации отдельной хромосомы. Молекулярные механизмы, которые контролируют количество актов инициации репликации в каждом сайте за один цикл деления клетки, называются контролем копийности. В бактериальных клетках помимо хромосомной ДНК часто содержатся плазмиды, которые представляют собой отдельные репликоны. У плазмид существуют свои механизмы контроля копийности: они могут обеспечивать синтез как всего одной копии плазмиды за клеточный цикл, так и тысяч копий.
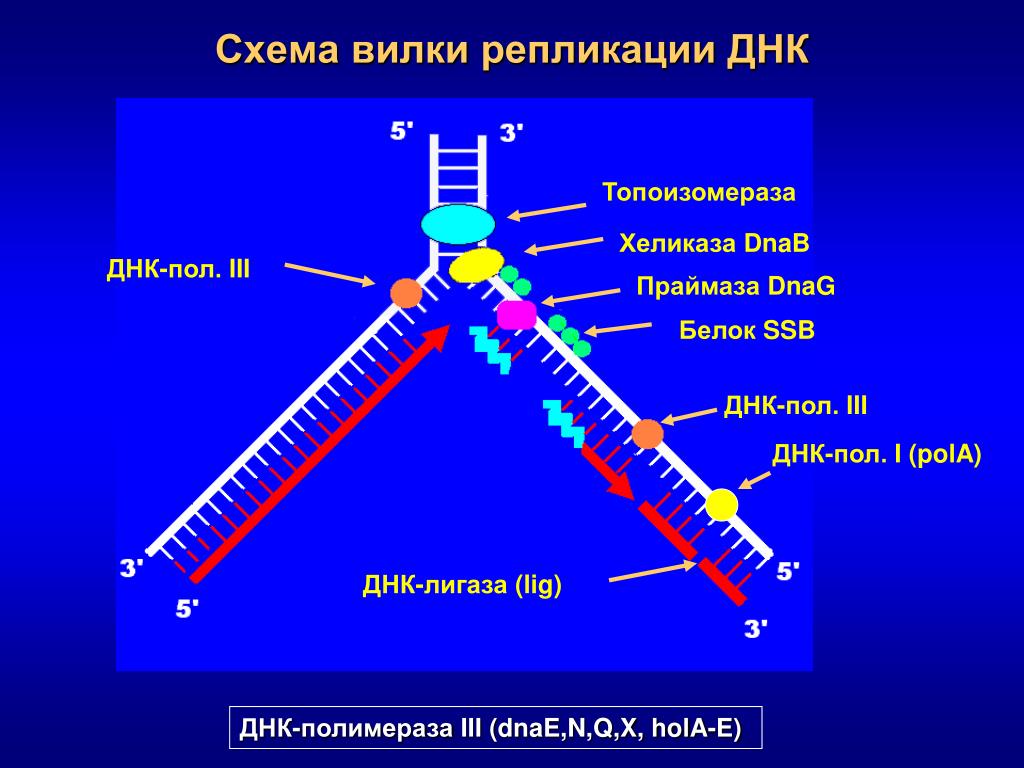
Репликация начинается в сайте инициации репликации с расплетания двойной спирали ДНК, при этом формируется репликационная вилка — место непосредственной репликации ДНК.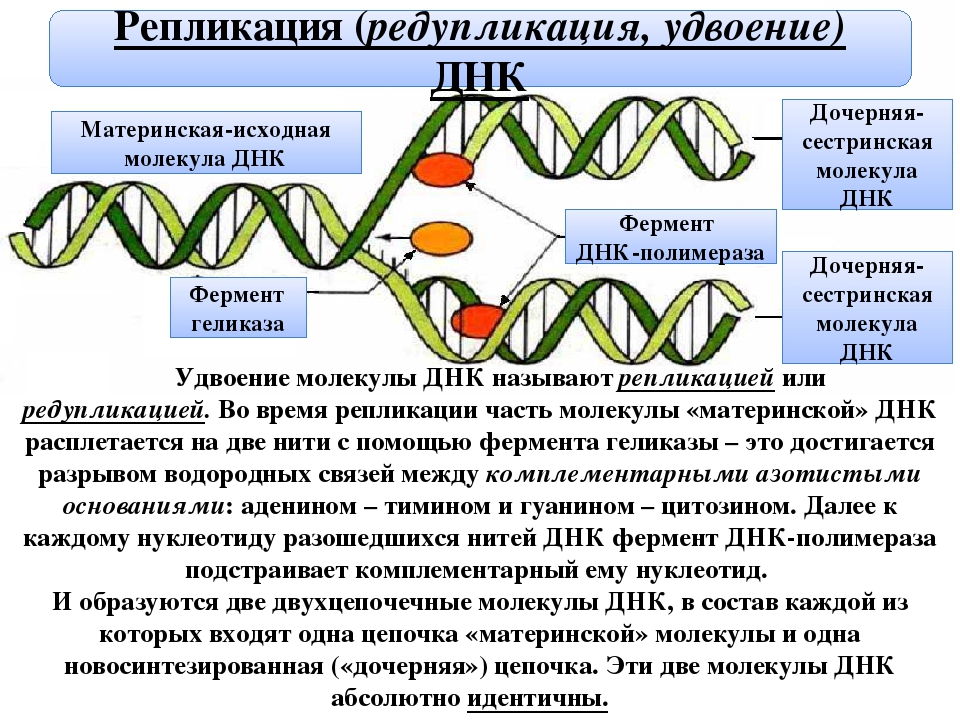 В каждом сайте может формироваться одна или две репликационные вилки в зависимости от того, является ли репликация одно- или двунаправленной. Более распространена двунаправленная репликация. Через некоторое время после начала репликации в электронный микроскоп можно наблюдать репликационный глазок — участок хромосомы, где ДНК уже реплицирована, окружённый более протяжёнными участками нереплицированной ДНК.
В каждом сайте может формироваться одна или две репликационные вилки в зависимости от того, является ли репликация одно- или двунаправленной. Более распространена двунаправленная репликация. Через некоторое время после начала репликации в электронный микроскоп можно наблюдать репликационный глазок — участок хромосомы, где ДНК уже реплицирована, окружённый более протяжёнными участками нереплицированной ДНК.
В репликационной вилке ДНК копирует крупный белковый комплекс реплисома, ключевым ферментом которого является ДНК-полимераза. Репликационная вилка движется со скоростью порядка 100 000 пар нуклеотидов в минуту у прокариот и 500 — 5000 — у эукариот.
Что такое репликация ДНК? | Факты
Репликация ДНК — это процесс, с помощью которого ДНК копирует себя во время деления клетки.
- Первым шагом в репликации ДНК является «расстегивание» двойной спиральной структуры молекулы ДНК.
- Это осуществляется ферментом под названием геликаза, который разрывает водородные связи, удерживающие вместе комплементарные основания ДНК (A с T, C с G).

- Разделение двух одиночных цепей ДНК создает Y-образную форму, называемую репликационной «вилкой».Две разделенные нити будут действовать как шаблоны для создания новых цепей ДНК.
- Одна из нитей ориентирована в направлении от 3 ’к 5’ (в сторону репликационной вилки), это ведущая нить. Другая нить ориентирована в направлении от 5 ’к 3’ (от вилки репликации), это отстающая нить. В результате их разной ориентации две цепи реплицируются по-разному:
Иллюстрация, показывающая репликацию ведущей и отстающей цепей ДНК.
Изображение предоставлено: Genome Research Limited
- Ведущая нить:
- Короткий фрагмент РНК, называемый праймером (продуцируемый ферментом, называемым праймазой), проходит и связывается с концом ведущей цепи. Праймер действует как отправная точка для синтеза ДНК.
- ДНК-полимераза связывается с ведущей цепью, а затем «идет» по ней, добавляя новые комплементарные нуклеотидные основания (A, C, G и T) к цепи ДНК в направлении от 5 ’к 3’.

- Этот вид репликации называется непрерывным.
- Отстающая цепь:
- Многочисленные праймеры РНК создаются ферментом праймазой и связываются в различных точках вдоль отстающей цепи.
- Куски ДНК, называемые фрагментами Окадзаки, затем добавляются к отстающей цепи также в направлении от 5 ’к 3’.
- Этот тип репликации называется прерывистой, так как фрагменты Окадзаки нужно будет объединить позже.
- После того, как все основания совпадают (A с T, C с G), фермент, называемый экзонуклеазой, удаляет праймер (ы).Затем пустоты, на которых находился праймер (ы), заполняются еще более комплементарными нуклеотидами.
- Новая цепь проверяется, чтобы убедиться в отсутствии ошибок в новой последовательности ДНК.
- Наконец, фермент под названием ДНК-лигаза запечатывает последовательность ДНК в две непрерывные двойные цепи.
- Результатом репликации ДНК являются две молекулы ДНК, состоящие из одной новой и одной старой цепочки нуклеотидов.
 Вот почему репликация ДНК описывается как полуконсервативная, половина цепи является частью исходной молекулы ДНК, половина — совершенно новой.
Вот почему репликация ДНК описывается как полуконсервативная, половина цепи является частью исходной молекулы ДНК, половина — совершенно новой. - После репликации новая ДНК автоматически сворачивается в двойную спираль.
Эта страница последний раз обновлялась 25.01.2016
Этапы и процесс репликации ДНК
Зачем реплицировать ДНК?
ДНК — это генетический материал, который определяет каждую клетку. Прежде чем клетка произведет дублирование и разделится на новые дочерние клетки посредством митоза или мейоза, биомолекулы и органеллы должны быть скопированы для распределения среди клеток.ДНК, обнаруженная в ядре, должна быть реплицирована, чтобы каждая новая клетка получала правильное количество хромосом. Процесс дублирования ДНК называется Репликация ДНК . Репликация состоит из нескольких этапов, в которых задействованы несколько белков, называемых ферментами репликации и РНК. В эукариотических клетках, таких как клетки животных и клетки растений, репликация ДНК происходит в S-фазе интерфазы во время клеточного цикла.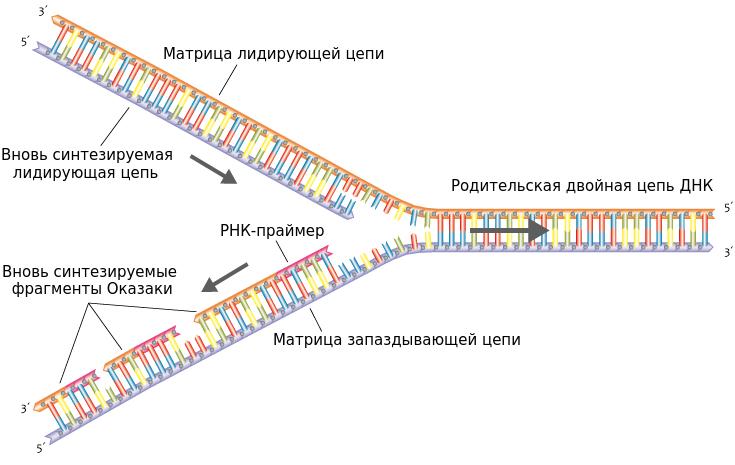 Процесс репликации ДНК жизненно важен для роста, восстановления и размножения клеток организмов.
Процесс репликации ДНК жизненно важен для роста, восстановления и размножения клеток организмов.
Ключевые выводы
- Дезоксирибонуклеиновая кислота, широко известная как ДНК, представляет собой нуклеиновую кислоту, которая состоит из трех основных компонентов: дезоксирибозного сахара, фосфата и азотистого основания.
- Поскольку ДНК содержит генетический материал для организма, важно, чтобы он копировался при делении клетки на дочерние клетки. Процесс копирования ДНК называется репликацией.
- Репликация включает получение идентичных спиралей ДНК из одной двухцепочечной молекулы ДНК.
- Ферменты жизненно важны для репликации ДНК, поскольку они катализируют очень важные шаги в этом процессе.
- Общий процесс репликации ДНК чрезвычайно важен как для роста клеток, так и для размножения организмов. Это также жизненно важно в процессе восстановления клеток.
Структура ДНК
ДНК или дезоксирибонуклеиновая кислота — это тип молекулы, известный как нуклеиновая кислота.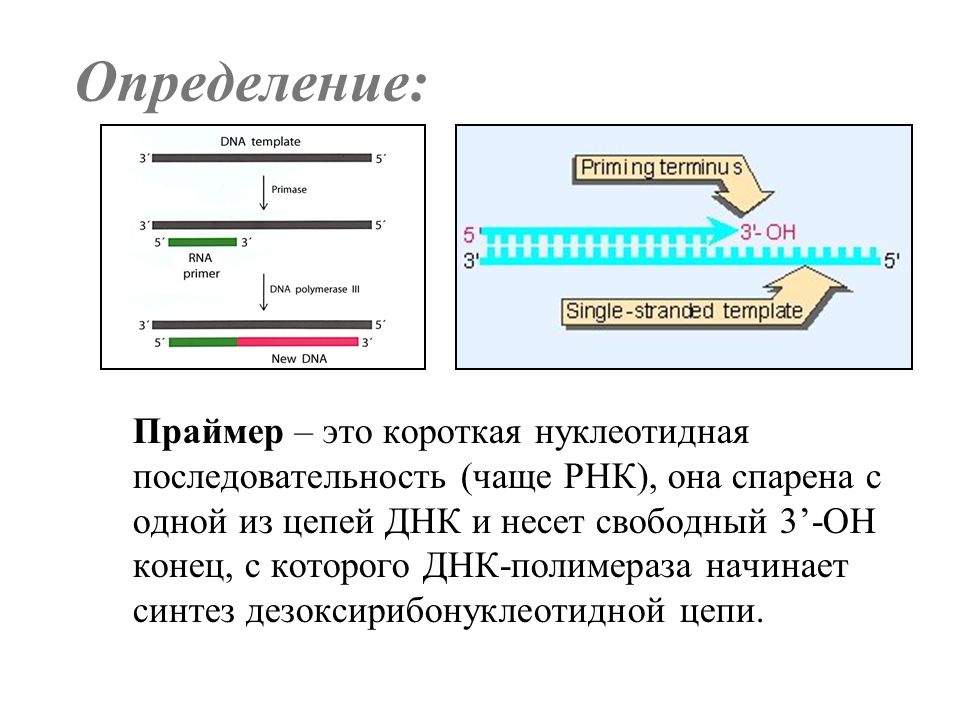 Он состоит из 5-углеродного дезоксирибозного сахара, фосфата и азотистого основания. Двухцепочечная ДНК состоит из двух спиральных цепей нуклеиновых кислот, скрученных в форму двойной спирали.Такое скручивание позволяет ДНК быть более компактной. Чтобы поместиться в ядре, ДНК упакована в плотно скрученные структуры, называемые хроматином. Хроматин конденсируется с образованием хромосом во время деления клетки. Перед репликацией ДНК хроматин разрыхляется, предоставляя механизмам репликации клеток доступ к цепям ДНК.
Он состоит из 5-углеродного дезоксирибозного сахара, фосфата и азотистого основания. Двухцепочечная ДНК состоит из двух спиральных цепей нуклеиновых кислот, скрученных в форму двойной спирали.Такое скручивание позволяет ДНК быть более компактной. Чтобы поместиться в ядре, ДНК упакована в плотно скрученные структуры, называемые хроматином. Хроматин конденсируется с образованием хромосом во время деления клетки. Перед репликацией ДНК хроматин разрыхляется, предоставляя механизмам репликации клеток доступ к цепям ДНК.
Подготовка к репликации
Библиотека научных фотографий / Getty Images
Шаг 1: Формирование репликационной вилки
Прежде чем ДНК сможет быть реплицирована, двухцепочечная молекула должна быть «разорвана» на две одноцепочечные.ДНК имеет четыре основания: аденин (A) , тимин (T) , цитозин (C) и гуанин (G) , которые образуют пары между двумя цепями. Аденин соединяется только с тимином, а цитозин связывается только с гуанином. Чтобы раскрутить ДНК, эти взаимодействия между парами оснований должны быть нарушены. Это выполняется ферментом, известным как ДНК геликаза . ДНК-геликаза разрушает водородные связи между парами оснований, разделяя нити в Y-образную форму, известную как репликационная вилка .Эта область будет шаблоном для начала репликации.
Чтобы раскрутить ДНК, эти взаимодействия между парами оснований должны быть нарушены. Это выполняется ферментом, известным как ДНК геликаза . ДНК-геликаза разрушает водородные связи между парами оснований, разделяя нити в Y-образную форму, известную как репликационная вилка .Эта область будет шаблоном для начала репликации.
ДНК направлена в обеих цепях, что обозначено 5 ‘и 3′ концом. Это обозначение означает, какая боковая группа присоединена к остову ДНК. К 5’-концу присоединена фосфатная (P) группа, а к 3′-концу присоединена гидроксильная (ОН) группа. Эта направленность важна для репликации, поскольку она прогрессирует только в направлении от 5 футов до 3 дюймов. Однако вилка репликации двунаправлена; одна нить ориентирована в направлении от 3 футов до 5 футов (ведущая нить) , а другая ориентирована от 5 футов до 3 футов (отстающая нить) .Таким образом, две стороны дублируются двумя разными процессами, чтобы учесть разницу в направлениях.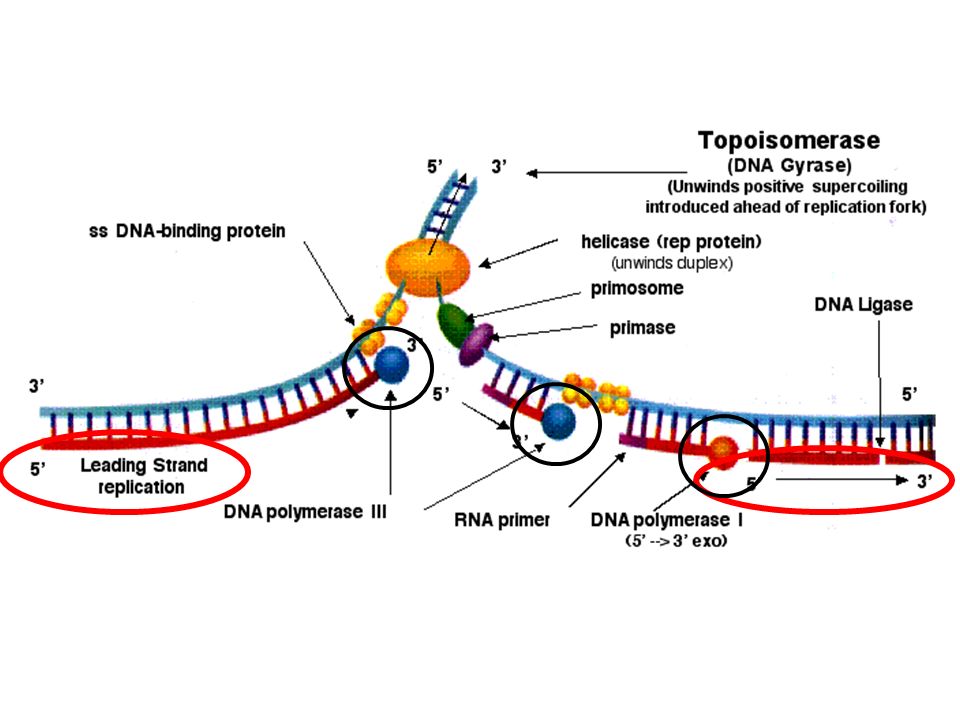
Начало репликации
Шаг 2: Привязка грунтовки
Ведущую нить проще всего воспроизвести. После разделения цепей ДНК короткий кусок РНК, называемый праймером , связывается с 3′-концом цепи. Праймер всегда связывается в качестве отправной точки для репликации. Праймеры генерируются ферментом ДНК-примазой .
Репликация ДНК: удлинение
ДНК-полимеразы (синий цвет) прикрепляются к ДНК и удлиняют новые цепи, добавляя нуклеотидные основания.UIG / Getty Images
Шаг 3: удлинение
Ферменты, известные как ДНК-полимеразы , ответственны за создание новой цепи посредством процесса, называемого удлинением. Существует пять различных известных типов ДНК-полимераз в бактериях и клетках человека. У бактерий, таких как E. coli, , полимераза III, является основным ферментом репликации, а полимеразы I, II, IV и V отвечают за проверку и исправление ошибок.ДНК-полимераза III связывается с цепью на участке праймера и начинает добавлять новые пары оснований, комплементарные цепи во время репликации.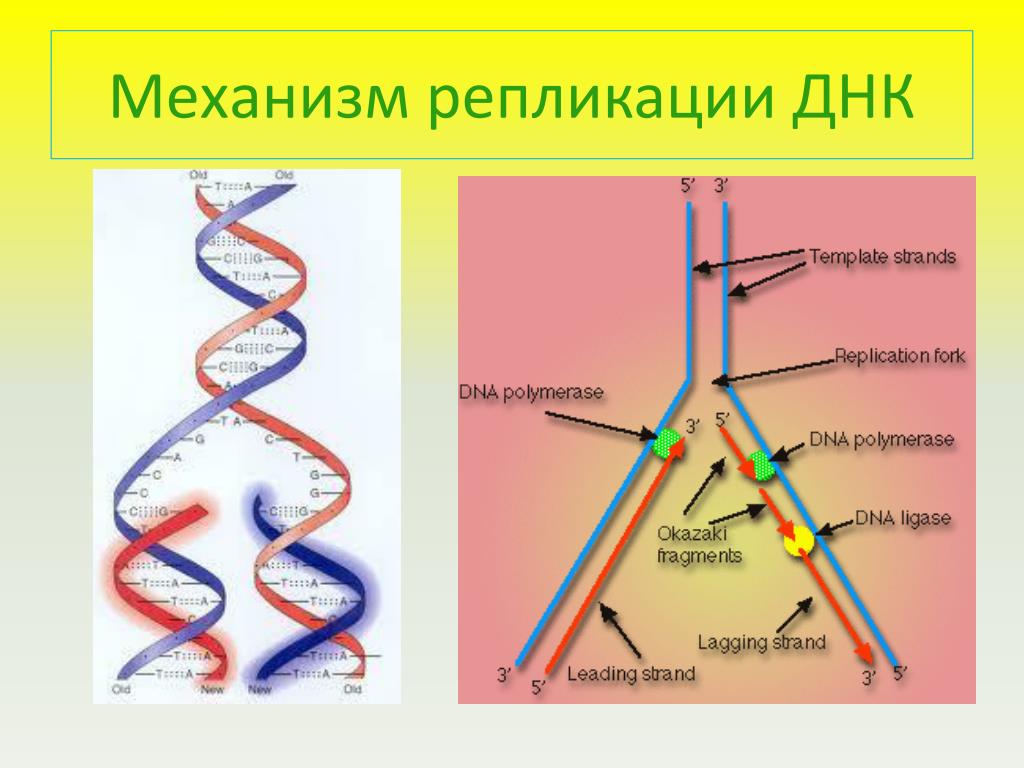 В эукариотических клетках полимеразы альфа, дельта и эпсилон являются первичными полимеразами, участвующими в репликации ДНК. Поскольку репликация происходит в направлении от 5 ‘до 3’ на ведущей цепи, вновь образованная цепь является непрерывной.
В эукариотических клетках полимеразы альфа, дельта и эпсилон являются первичными полимеразами, участвующими в репликации ДНК. Поскольку репликация происходит в направлении от 5 ‘до 3’ на ведущей цепи, вновь образованная цепь является непрерывной.
Отстающая цепь начинает репликацию путем связывания с несколькими праймерами. Каждый праймер состоит всего из нескольких оснований.Затем ДНК-полимераза добавляет к цепи между праймерами фрагменты ДНК, называемые фрагментами Окадзаки . Этот процесс репликации прерывистый, так как вновь созданные фрагменты разъединены.
Шаг 4: Прекращение действия
После образования как непрерывных, так и прерывистых цепей фермент под названием экзонуклеаза удаляет все праймеры РНК из исходных цепей. Затем эти праймеры заменяют соответствующими основаниями. Другая экзонуклеаза «корректирует» вновь образованную ДНК, чтобы проверить, удалить и заменить любые ошибки.Другой фермент, называемый ДНК-лигаза , объединяет фрагменты Окадзаки вместе, образуя единую цепь.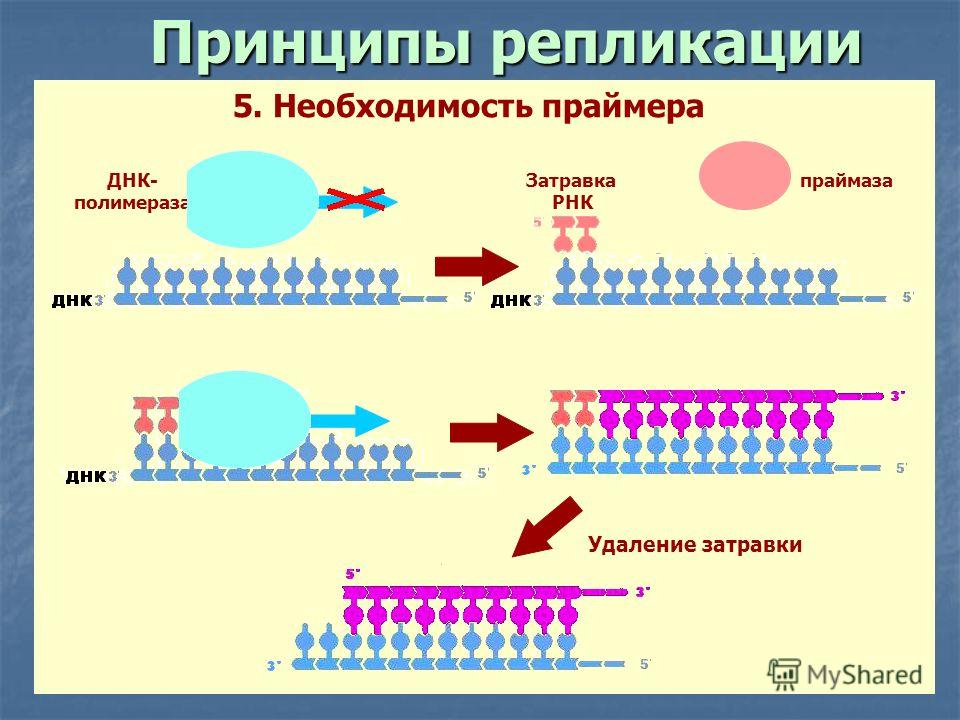 Концы линейной ДНК представляют проблему, поскольку ДНК-полимераза может добавлять нуклеотиды только в направлении от 5 ‘к 3’. Концы родительских цепей состоят из повторяющихся последовательностей ДНК, называемых теломерами. Теломеры действуют как защитные колпачки на концах хромосом, предотвращая слияние соседних хромосом. Особый тип фермента ДНК-полимеразы под названием теломераза катализирует синтез теломерных последовательностей на концах ДНК.После завершения родительская цепь и ее комплементарная цепь ДНК сворачиваются в знакомую форму двойной спирали. В конце концов, при репликации образуются две молекулы ДНК, каждая с одной цепью исходной молекулы и одной новой цепью.
Концы линейной ДНК представляют проблему, поскольку ДНК-полимераза может добавлять нуклеотиды только в направлении от 5 ‘к 3’. Концы родительских цепей состоят из повторяющихся последовательностей ДНК, называемых теломерами. Теломеры действуют как защитные колпачки на концах хромосом, предотвращая слияние соседних хромосом. Особый тип фермента ДНК-полимеразы под названием теломераза катализирует синтез теломерных последовательностей на концах ДНК.После завершения родительская цепь и ее комплементарная цепь ДНК сворачиваются в знакомую форму двойной спирали. В конце концов, при репликации образуются две молекулы ДНК, каждая с одной цепью исходной молекулы и одной новой цепью.
Ферменты репликации
Молекула ДНК-полимеразы.Cultura / Getty Images
Репликация ДНК не могла бы происходить без ферментов, которые катализируют различные стадии процесса. Ферменты, которые участвуют в процессе репликации эукариотической ДНК, включают:
- ДНК-геликаза — раскручивает и разделяет двухцепочечную ДНК по мере ее движения вдоль ДНК.
 Он образует репликационную вилку, разрывая водородные связи между парами нуклеотидов в ДНК.
Он образует репликационную вилку, разрывая водородные связи между парами нуклеотидов в ДНК. - ДНК-примаза — тип РНК-полимеразы, которая генерирует РНК-праймеры. Праймеры — это короткие молекулы РНК, которые действуют как матрицы для начальной точки репликации ДНК.
- ДНК-полимеразы — синтезируют новые молекулы ДНК путем добавления нуклеотидов к ведущим и отстающим цепям ДНК.
- Топоизомераза или ДНК-гираза — раскручивает и перематывает цепи ДНК, чтобы предотвратить запутывание или сверхспирали ДНК.
- Экзонуклеазы — группа ферментов, удаляющих нуклеотидные основания с конца цепи ДНК.
- ДНК-лигаза — соединяет фрагменты ДНК вместе, образуя фосфодиэфирные связи между нуклеотидами.
Сводка репликации ДНК
Репликация ДНК.Фрэнсис Лерой / Getty Images
Репликация ДНК — это производство идентичных спиралей ДНК из одной двухцепочечной молекулы ДНК. Каждая молекула состоит из цепочки исходной молекулы и вновь образованной цепи.Перед репликацией ДНК разматывается, и нити разделяются. Формируется репликационная вилка, которая служит шаблоном для репликации. Праймеры связываются с ДНК, и ДНК-полимеразы добавляют новые нуклеотидные последовательности в направлении от 5 ‘к 3’.
Каждая молекула состоит из цепочки исходной молекулы и вновь образованной цепи.Перед репликацией ДНК разматывается, и нити разделяются. Формируется репликационная вилка, которая служит шаблоном для репликации. Праймеры связываются с ДНК, и ДНК-полимеразы добавляют новые нуклеотидные последовательности в направлении от 5 ‘к 3’.
Это добавление непрерывно в ведущей нити и фрагментировано в отстающей нити. Как только удлинение цепей ДНК завершено, цепи проверяются на наличие ошибок, производится ремонт и к концам ДНК добавляются теломерные последовательности.
Источники
- Рис, Джейн Б., и Нил А. Кэмпбелл. Биология Кэмпбелла . Бенджамин Каммингс, 2011.
Смотри: Что такое двоичное деление?
репликации ДНК | Безграничная биология
Основы репликации ДНК
Репликация ДНК использует полуконсервативный метод, который приводит к получению двухцепочечной ДНК с одной родительской цепью и новой дочерней цепью.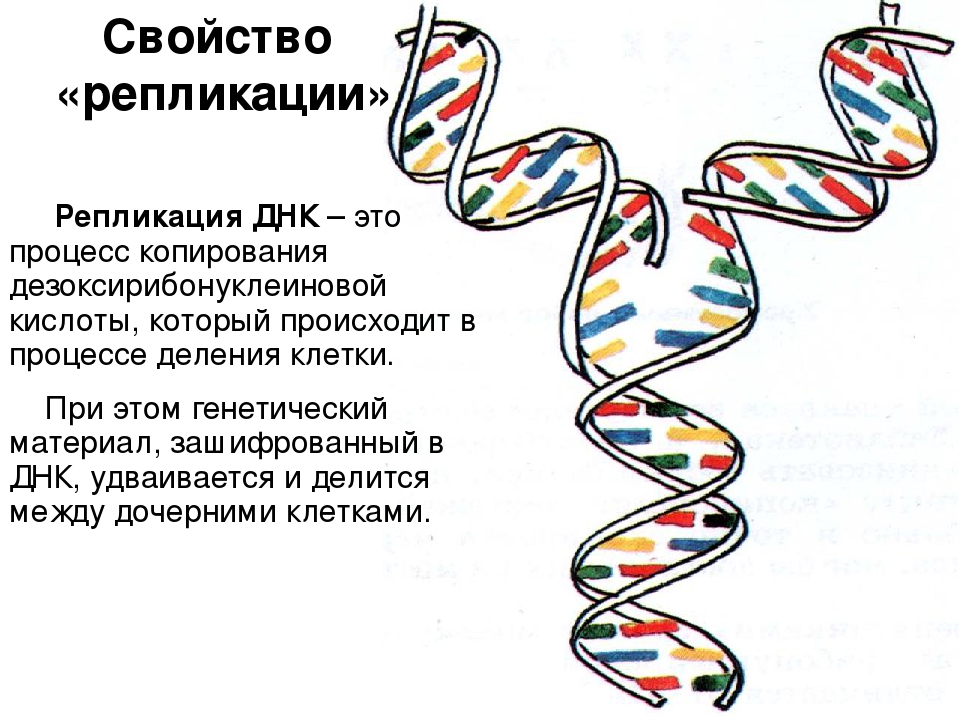
Цели обучения
Объясните, как эксперимент Мезельсона и Шталь окончательно установил, что репликация ДНК полуконсервативна.
Основные выводы
Ключевые моменты
- Было предложено три модели репликации ДНК: консервативная, полуконсервативная и дисперсионная.
- Консервативный метод репликации предполагает, что родительская ДНК остается вместе, и вновь образованные дочерние цепи также находятся вместе.
- Полуконсервативный метод репликации предполагает, что две родительские цепи ДНК служат в качестве матрицы для новой ДНК, а после репликации каждая двухцепочечная ДНК содержит одну цепь родительской ДНК и одну новую (дочернюю) цепь.
- Дисперсионный метод репликации предполагает, что после репликации две дочерние ДНК имеют чередующиеся сегменты как родительской, так и вновь синтезированной ДНК, вкрапленные на обеих цепях.
- Meselson и Stahl, используя ДНК E. coli , полученную с двумя истопами азота ( 14 N и 15 N), и центрифугирование в градиенте плотности, определили, что ДНК реплицируется с помощью полуконсервативного метода репликации.

Ключевые термины
- Репликация ДНК : биологический процесс, происходящий во всех живых организмах, который является основой биологической наследственности
- изотоп : любая из двух или более форм элемента, в которых атомы имеют одинаковое количество протонов, но разное количество нейтронов в их ядрах
Основы репликации ДНК
Открытие Уотсона и Крика, что ДНК представляет собой двухцепочечную двойную спираль, дало намек на то, как реплицируется ДНК.Во время деления клетки каждая молекула ДНК должна быть идеально скопирована, чтобы гарантировать перемещение идентичных молекул ДНК к каждой из двух дочерних клеток. Двухцепочечная структура ДНК предполагает, что две цепи могут разделяться во время репликации, причем каждая цепь служит шаблоном, из которого копируется новая комплементарная цепь для каждой, генерируя две двухцепочечные молекулы из одной.
Модели репликации
При такой схеме возможны три модели репликации: консервативная, полуконсервативная и дисперсионная.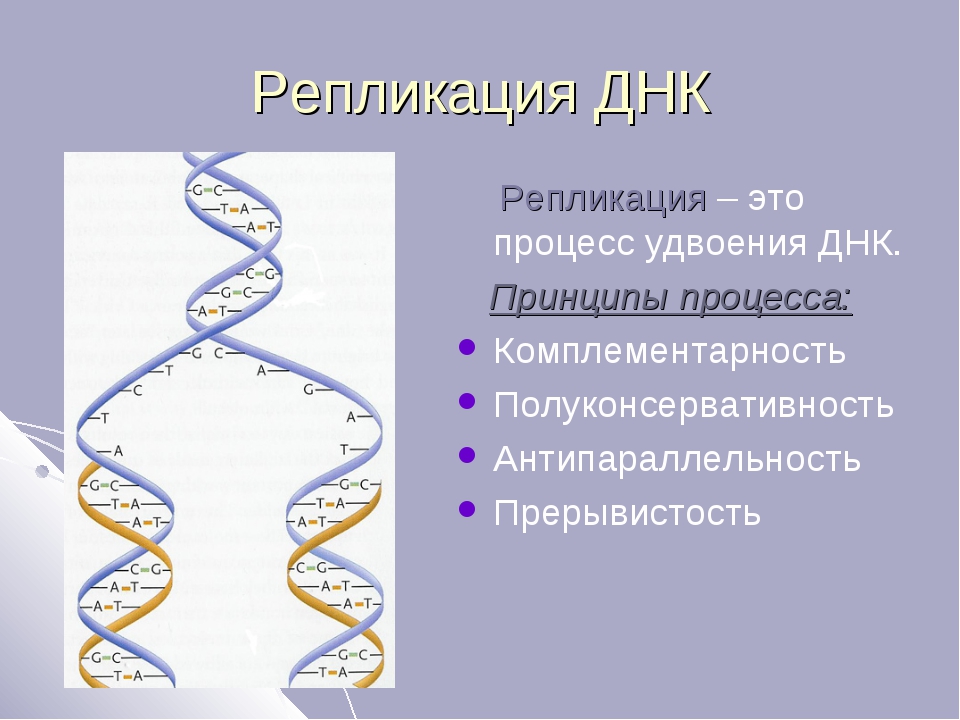 При консервативной репликации две исходные цепи ДНК, известные как родительские цепи, будут повторно образовывать пары оснований друг с другом после использования в качестве матриц для синтеза новых цепей; и две вновь синтезированные нити, известные как дочерние нити, также будут спариваться друг с другом по основанию; одна из двух молекул ДНК после репликации будет «полностью старой», а другая — «совершенно новой». При полуконсервативной репликации каждая из двух родительских цепей ДНК будет действовать как матрица для новых цепей ДНК, которые должны быть синтезированы, но после репликации каждая родительская цепочка ДНК будет спариваться по основанию с комплементарной вновь синтезированной цепью, только что синтезированной, и обе будут двойными. Нитевые ДНК могут включать одну родительскую или «старую» цепь и одну дочернюю или «новую» цепь.При дисперсионной репликации после репликации обе копии новой ДНК каким-то образом будут иметь чередующиеся сегменты родительской ДНК и вновь синтезированной ДНК на каждой из своих двух цепей.
При консервативной репликации две исходные цепи ДНК, известные как родительские цепи, будут повторно образовывать пары оснований друг с другом после использования в качестве матриц для синтеза новых цепей; и две вновь синтезированные нити, известные как дочерние нити, также будут спариваться друг с другом по основанию; одна из двух молекул ДНК после репликации будет «полностью старой», а другая — «совершенно новой». При полуконсервативной репликации каждая из двух родительских цепей ДНК будет действовать как матрица для новых цепей ДНК, которые должны быть синтезированы, но после репликации каждая родительская цепочка ДНК будет спариваться по основанию с комплементарной вновь синтезированной цепью, только что синтезированной, и обе будут двойными. Нитевые ДНК могут включать одну родительскую или «старую» цепь и одну дочернюю или «новую» цепь.При дисперсионной репликации после репликации обе копии новой ДНК каким-то образом будут иметь чередующиеся сегменты родительской ДНК и вновь синтезированной ДНК на каждой из своих двух цепей.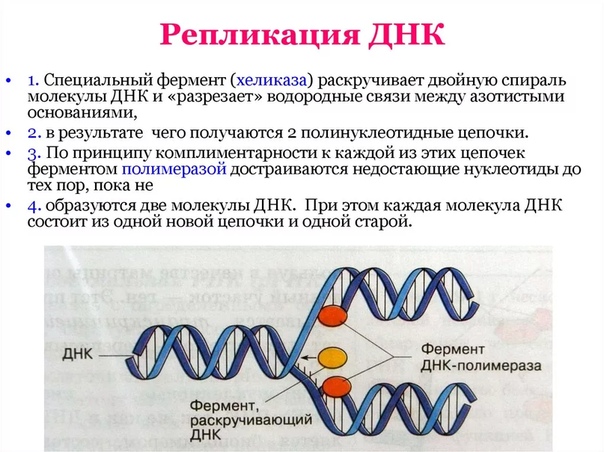
Предлагаемые модели репликации ДНК : Три предложенных модели репликации ДНК. Серый цвет указывает на исходные цепи или сегменты родительской ДНК, а синий цвет указывает на вновь синтезированные цепи или сегменты дочерней ДНК.
Чтобы определить, какая модель репликации является точной, в 1958 году двумя исследователями: Мэтью Мезельсоном и Франклином Шталом был проведен плодотворный эксперимент.
Meselson и Stahl
Мезельсон и Шталь интересовались тем, как реплицируется ДНК. Они выращивали E. coli в течение нескольких поколений в среде, содержащей «тяжелый» изотоп азота ( 15 N), который включен в азотистые основания и, в конечном итоге, в ДНК. Затем культуру E. coli переносили в среду, содержащую обычный «легкий» изотоп азота ( 14 N), и позволяли расти в течение одного поколения. Клетки собирали и выделяли ДНК.ДНК центрифугировали на высоких скоростях в ультрацентрифуге в пробирке, в которой был установлен градиент плотности хлорида цезия. Некоторым клеткам давали возможность вырасти еще на один жизненный цикл в 14 N и снова вращали.
Некоторым клеткам давали возможность вырасти еще на один жизненный цикл в 14 N и снова вращали.
Meselson and Stahl : Meselson и Stahl экспериментировали с E. coli, выращенной сначала в тяжелом азоте ( 15 N), затем в более низком азоте ( 14 N). ДНК, выращенная в 15 N (красная полоса), тяжелее чем ДНК, выращенная в 14 N (оранжевая полоса) и осаждающая до более низкого уровня градиента плотности хлорида цезия в ультрацентрифуге.Когда ДНК, выращенная в 15 N, переключается на среду, содержащую 14 N, после одного раунда деления клетки ДНК оседает на полпути между уровнями 15 N и 14 N, показывая, что теперь она содержит пятьдесят процентов 14 N и пятьдесят процентов 15 N. При последующих делениях клеток все большее количество ДНК содержит только 14 N. Эти данные поддерживают полуконсервативную модель репликации.
Во время ультрацентрифугирования в градиенте плотности ДНК загружали в градиент (Мезельсон и Шталь использовали градиент соли хлорида цезия, хотя для создания градиента также можно использовать другие материалы, такие как сахароза) и вращали с высокой скоростью от 50 000 до 60 000.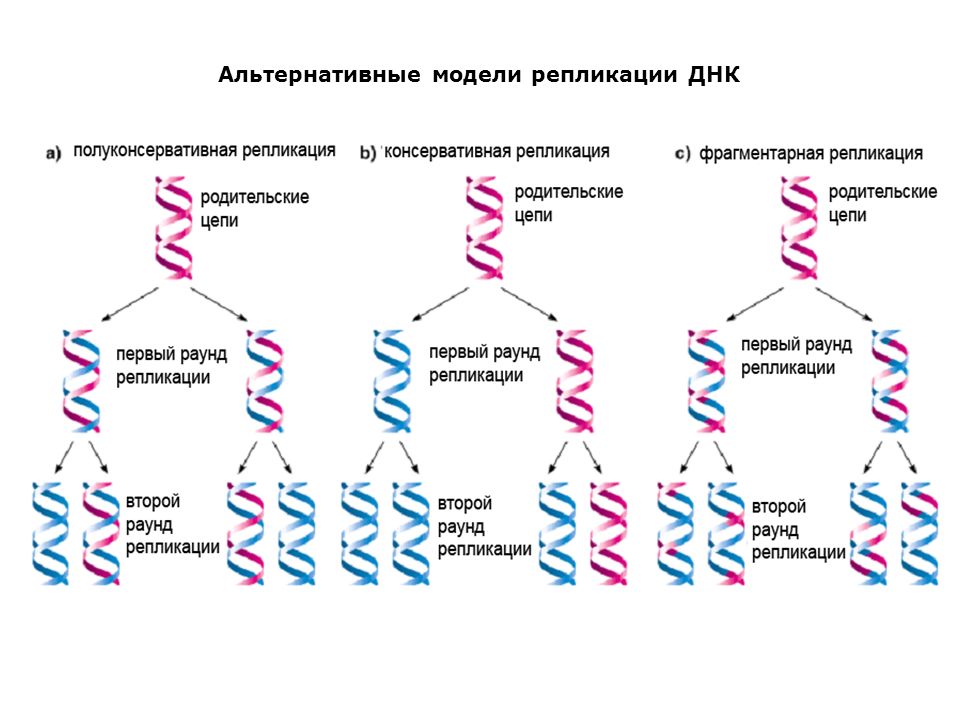 об / мин.В ультрацентрифужной пробирке соль хлорида цезия создавала градиент плотности, причем раствор хлорида цезия был тем плотнее, чем дальше вы спускались по трубке. В этих условиях во время вращения ДНК протягивалась вниз по ультрацентрифужной пробирке под действием центробежной силы до тех пор, пока она не достигла точки в градиенте соли, где плотность молекул ДНК соответствовала плотности окружающего солевого раствора. В этот момент молекулы перестали оседать и образовали стабильную полосу. Глядя на относительное положение полос молекул, движущихся по одним и тем же градиентам, вы можете определить относительные плотности различных молекул.Молекулы, образующие самые низкие полосы, имеют наибольшую плотность.
об / мин.В ультрацентрифужной пробирке соль хлорида цезия создавала градиент плотности, причем раствор хлорида цезия был тем плотнее, чем дальше вы спускались по трубке. В этих условиях во время вращения ДНК протягивалась вниз по ультрацентрифужной пробирке под действием центробежной силы до тех пор, пока она не достигла точки в градиенте соли, где плотность молекул ДНК соответствовала плотности окружающего солевого раствора. В этот момент молекулы перестали оседать и образовали стабильную полосу. Глядя на относительное положение полос молекул, движущихся по одним и тем же градиентам, вы можете определить относительные плотности различных молекул.Молекулы, образующие самые низкие полосы, имеют наибольшую плотность.
из клеток, выращенных исключительно в 15 N, давала более низкую полосу, чем ДНК из клеток, выращенных исключительно в 14 N. Таким образом, ДНК, выращенная в 15 N, имела более высокую плотность, как и следовало ожидать от молекулы с более тяжелым изотоп азота входит в его азотистые основания.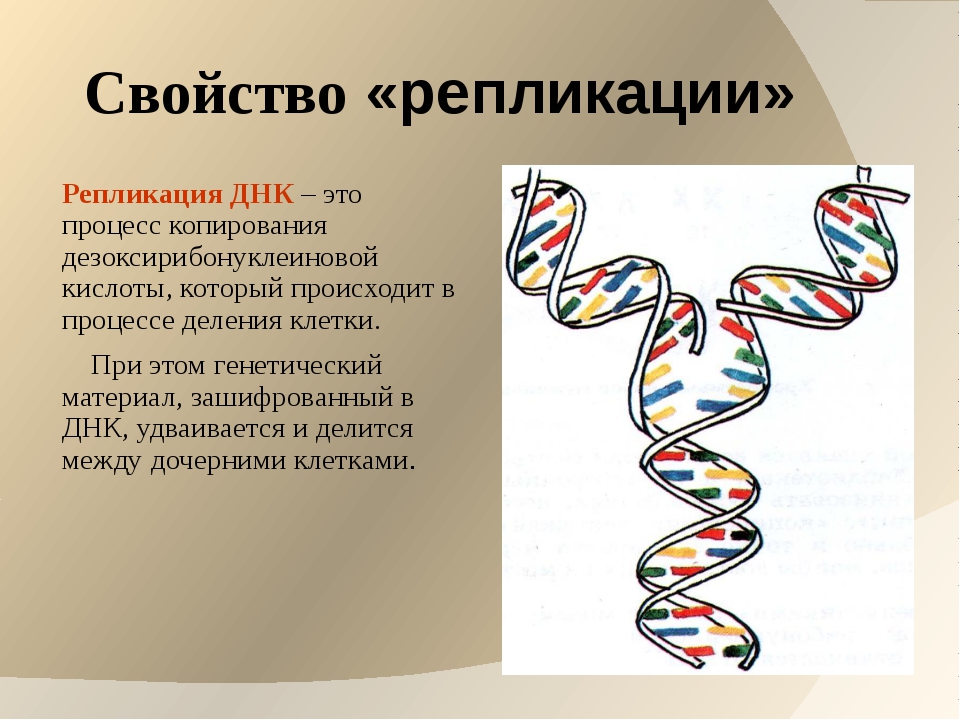 Мезельсон и Шталь отметили, что после одного поколения роста в 14 N (после того, как клетки были сдвинуты с 15 N), молекулы ДНК продуцировали только одно полосное промежуточное соединение в положении между ДНК клеток, выращенных исключительно в 15 N. и ДНК клеток, выращенных исключительно в 14 N.Это предполагает либо полуконсервативный, либо дисперсионный режим репликации. Консервативная репликация привела бы к двум полосам; одна представляет собой родительскую ДНК с исключительно 15 N в ее азотистых основаниях, а другая представляет собой дочернюю ДНК с исключительно 14 N в ее азотистых основаниях. Фактически наблюдаемая единственная полоса указывает на то, что все молекулы ДНК содержат равные количества 15 N и 14 N.
Мезельсон и Шталь отметили, что после одного поколения роста в 14 N (после того, как клетки были сдвинуты с 15 N), молекулы ДНК продуцировали только одно полосное промежуточное соединение в положении между ДНК клеток, выращенных исключительно в 15 N. и ДНК клеток, выращенных исключительно в 14 N.Это предполагает либо полуконсервативный, либо дисперсионный режим репликации. Консервативная репликация привела бы к двум полосам; одна представляет собой родительскую ДНК с исключительно 15 N в ее азотистых основаниях, а другая представляет собой дочернюю ДНК с исключительно 14 N в ее азотистых основаниях. Фактически наблюдаемая единственная полоса указывает на то, что все молекулы ДНК содержат равные количества 15 N и 14 N.
ДНК, полученная из клеток, выращенных в двух поколениях в 14 N, образовала две полосы: одна полоса ДНК находилась в промежуточном положении между 15 N и 14 N, а другая соответствовала полосе исключительно 14 N.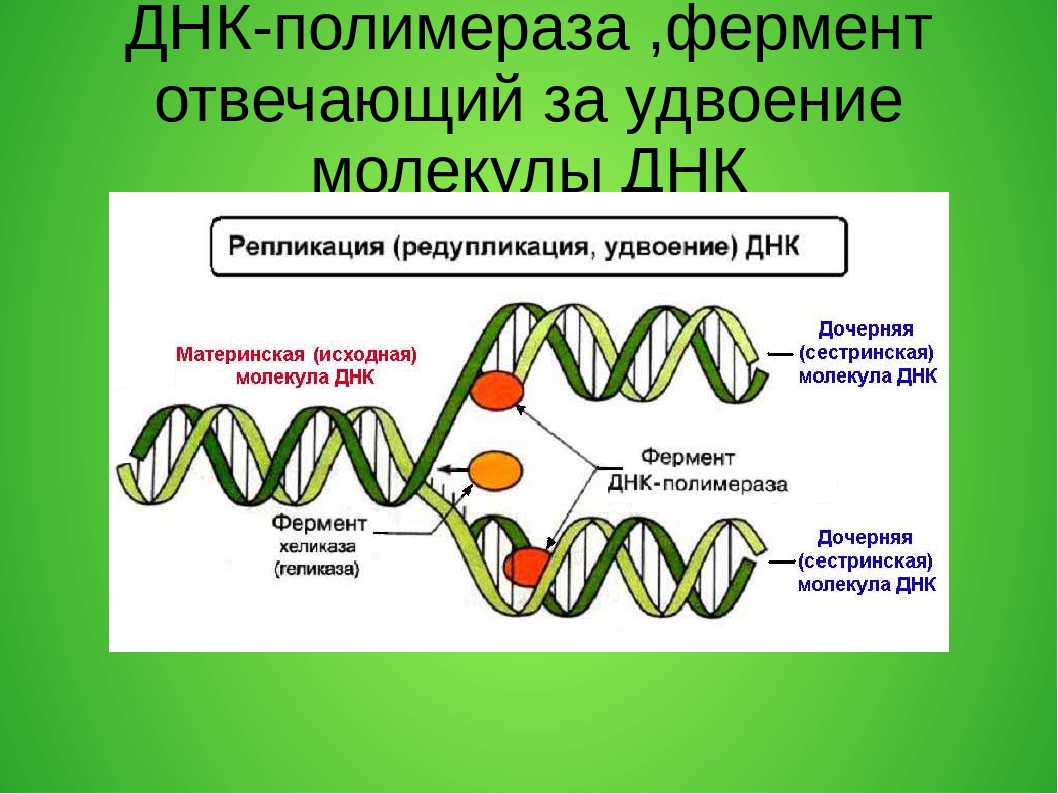 ДНК.Эти результаты можно объяснить только в том случае, если ДНК реплицируется полуконсервативным образом. Дисперсионная репликация привела бы к исключительно одной полосе в каждом новом поколении, при этом полоса медленно приближалась бы к высоте полосы ДНК 14 N. Следовательно, можно исключить и дисперсионную репликацию.
ДНК.Эти результаты можно объяснить только в том случае, если ДНК реплицируется полуконсервативным образом. Дисперсионная репликация привела бы к исключительно одной полосе в каждом новом поколении, при этом полоса медленно приближалась бы к высоте полосы ДНК 14 N. Следовательно, можно исключить и дисперсионную репликацию.
Результаты Meselson и Stahl установили, что во время репликации ДНК каждая из двух цепей, составляющих двойную спираль, служит шаблоном, из которого синтезируются новые цепи.Новая нить будет дополнять родительскую или «старую» нить, а новая нить останется связанной со старой нитью. Таким образом, каждая «дочерняя» ДНК фактически состоит из одной «старой» цепи ДНК и одной недавно синтезированной цепи. Когда образуются две дочерние копии ДНК, они имеют идентичные друг другу последовательности и последовательности, идентичные исходной родительской ДНК, и две дочерние ДНК делятся поровну на две дочерние клетки, производя дочерние клетки, которые генетически идентичны друг другу и генетически идентичен родительской клетке.
Репликация ДНК у прокариот
Прокариотическая ДНК реплицируется ДНК-полимеразой III в направлении от 5 ‘к 3’ со скоростью 1000 нуклеотидов в секунду.
Цели обучения
Объясните функции ферментов, участвующих в репликации прокариотической ДНК
Основные выводы
Ключевые моменты
- Геликаза разделяет ДНК с образованием репликационной вилки в точке начала репликации, где начинается репликация ДНК.
- Вилки репликации расширяются в двух направлениях по мере продолжения репликации.
- фрагментов Окадзаки образуются на отстающей цепи, в то время как ведущая цепь непрерывно реплицируется.
- ДНК-лигаза закрывает промежутки между фрагментами Окадзаки.
- Примаза синтезирует праймер РНК со свободным 3′-ОН, который ДНК-полимераза III использует для синтеза дочерних цепей.
Ключевые термины
- Репликация ДНК : биологический процесс, происходящий во всех живых организмах, который является основой биологической наследственности
- геликаза : фермент, раскручивающий спираль ДНК перед механизмом репликации
- точка начала репликации : конкретная последовательность в геноме, в которой начинается репликация
Репликация ДНК у прокариот
В репликации ДНК задействовано большое количество белков и ферментов, каждый из которых играет важную роль в процессе. Одним из ключевых игроков является фермент ДНК-полимераза, который добавляет нуклеотиды один за другим к растущей цепи ДНК, комплементарной цепи матрицы. Добавление нуклеотидов требует энергии; эта энергия получена от нуклеотидов, к которым присоединены три фосфата, подобно АТФ, у которого есть три присоединенные фосфатные группы. Когда связь между фосфатами разрывается, высвобождаемая энергия используется для образования фосфодиэфирной связи между поступающим нуклеотидом и растущей цепью.У прокариот известны три основных типа полимераз: ДНК pol I, ДНК pol II и ДНК pol III. ДНК pol III — это фермент, необходимый для синтеза ДНК; ДНК pol I и ДНК pol II в первую очередь необходимы для восстановления.
Одним из ключевых игроков является фермент ДНК-полимераза, который добавляет нуклеотиды один за другим к растущей цепи ДНК, комплементарной цепи матрицы. Добавление нуклеотидов требует энергии; эта энергия получена от нуклеотидов, к которым присоединены три фосфата, подобно АТФ, у которого есть три присоединенные фосфатные группы. Когда связь между фосфатами разрывается, высвобождаемая энергия используется для образования фосфодиэфирной связи между поступающим нуклеотидом и растущей цепью.У прокариот известны три основных типа полимераз: ДНК pol I, ДНК pol II и ДНК pol III. ДНК pol III — это фермент, необходимый для синтеза ДНК; ДНК pol I и ДНК pol II в первую очередь необходимы для восстановления.
Существуют определенные нуклеотидные последовательности, называемые точками репликации, где начинается репликация. У E. coli , который имеет единственную точку начала репликации на одной хромосоме (как и большинство прокариот), он имеет длину приблизительно 245 пар оснований и богат последовательностями AT. Источник репликации распознается некоторыми белками, которые связываются с этим сайтом. Фермент под названием геликаза раскручивает ДНК, разрывая водородные связи между парами азотистых оснований. Для этого процесса требуется гидролиз АТФ. Когда ДНК открывается, образуются Y-образные структуры, называемые репликационными вилками. Две репликационные вилки в источнике репликации расширяются в двух направлениях по мере продолжения репликации. Одноцепочечные связывающие белки покрывают нити ДНК возле репликационной вилки, чтобы предотвратить повторное скручивание однонитевой ДНК в двойную спираль.ДНК-полимераза способна добавлять нуклеотиды только в направлении от 5 ‘к 3′ (новая цепь ДНК может быть удлинена только в этом направлении). Также требуется свободная 3′-OH группа, к которой он может присоединять нуклеотиды, образуя фосфодиэфирную связь между 3′-OH концом и 5′-фосфатом следующего нуклеотида. Это означает, что он не может добавлять нуклеотиды, если свободная 3’-ОН группа недоступна.
Источник репликации распознается некоторыми белками, которые связываются с этим сайтом. Фермент под названием геликаза раскручивает ДНК, разрывая водородные связи между парами азотистых оснований. Для этого процесса требуется гидролиз АТФ. Когда ДНК открывается, образуются Y-образные структуры, называемые репликационными вилками. Две репликационные вилки в источнике репликации расширяются в двух направлениях по мере продолжения репликации. Одноцепочечные связывающие белки покрывают нити ДНК возле репликационной вилки, чтобы предотвратить повторное скручивание однонитевой ДНК в двойную спираль.ДНК-полимераза способна добавлять нуклеотиды только в направлении от 5 ‘к 3′ (новая цепь ДНК может быть удлинена только в этом направлении). Также требуется свободная 3′-OH группа, к которой он может присоединять нуклеотиды, образуя фосфодиэфирную связь между 3′-OH концом и 5′-фосфатом следующего нуклеотида. Это означает, что он не может добавлять нуклеотиды, если свободная 3’-ОН группа недоступна. Другой фермент, РНК-примаза, синтезирует праймер РНК, который имеет длину от пяти до десяти нуклеотидов и комплементарен ДНК, инициируя синтез ДНК.Праймер обеспечивает свободный 3′-ОН конец для начала репликации. Затем ДНК-полимераза удлиняет этот праймер РНК, добавляя один за другим нуклеотиды, комплементарные цепи матрицы.
Другой фермент, РНК-примаза, синтезирует праймер РНК, который имеет длину от пяти до десяти нуклеотидов и комплементарен ДНК, инициируя синтез ДНК.Праймер обеспечивает свободный 3′-ОН конец для начала репликации. Затем ДНК-полимераза удлиняет этот праймер РНК, добавляя один за другим нуклеотиды, комплементарные цепи матрицы.
Репликация ДНК у прокариот : Репликационная вилка образуется, когда геликаза разделяет цепи ДНК в ориджине репликации. ДНК имеет тенденцию становиться более свернутой перед репликационной вилкой. Топоизомераза разрывает и реформирует фосфатный остов ДНК перед вилкой репликации, тем самым снимая давление, возникающее в результате этой суперспирализации.Одноцепочечные связывающие белки связываются с одноцепочечной ДНК, чтобы предотвратить повторное формирование спирали. Примаза синтезирует праймер РНК. ДНК-полимераза III использует этот праймер для синтеза дочерней цепи ДНК. На ведущей цепи ДНК синтезируется непрерывно, тогда как на отстающей цепи ДНК синтезируется короткими отрезками, называемыми фрагментами Окадзаки. ДНК-полимераза I заменяет праймер РНК ДНК. ДНК-лигаза закрывает промежутки между фрагментами Окадзаки, объединяя фрагменты в единую молекулу ДНК.
ДНК-полимераза I заменяет праймер РНК ДНК. ДНК-лигаза закрывает промежутки между фрагментами Окадзаки, объединяя фрагменты в единую молекулу ДНК.
Репликационная вилка перемещается со скоростью 1000 нуклеотидов в секунду. ДНК-полимераза может распространяться только в направлении от 5 ‘к 3’, что создает небольшую проблему на вилке репликации. Как мы знаем, двойная спираль ДНК антипараллельна; то есть одна нить находится в направлении от 5 ‘до 3’, а другая — в направлении от 3 ‘до 5’. Одна цепь (ведущая цепь), комплементарная родительской цепи ДНК от 3 ‘до 5’, непрерывно синтезируется в направлении репликационной вилки, поскольку полимераза может добавлять нуклеотиды в этом направлении.Другая цепь (отстающая цепь), комплементарная родительской ДНК от 5 ‘до 3’, удлиняется от репликационной вилки небольшими фрагментами, известными как фрагменты Окадзаки, каждый из которых требует праймера для начала синтеза. Фрагменты Окадзаки названы в честь японского ученого, впервые обнаружившего их.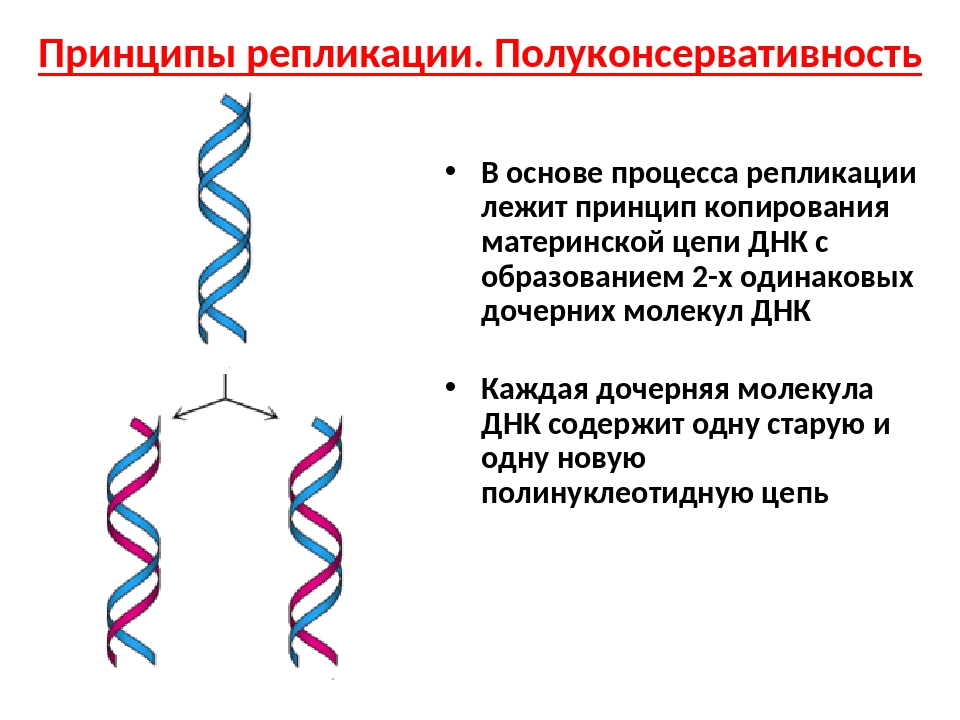
Ведущую цепь можно удлинить одним праймером, тогда как отстающей цепи нужен новый праймер для каждого из коротких фрагментов Окадзаки. Общее направление отстающей нити будет от 3 ‘до 5’, а направление ведущей нити будет от 5 ‘до 3’.Скользящий зажим (белок в форме кольца, который связывается с ДНК) удерживает ДНК-полимеразу на месте, пока она продолжает добавлять нуклеотиды. Топоизомераза предотвращает закручивание двойной спирали ДНК перед репликационной вилкой, когда ДНК открывается; он делает это, создавая временные разрывы в спирали ДНК, а затем снова запечатывая ее. По мере синтеза праймеры РНК заменяются ДНК. Праймеры удаляются экзонуклеазной активностью ДНК pol I, в то время как пробелы заполняются дезоксирибонуклеотидами.Разрывы, которые остаются между вновь синтезированной ДНК (которая заменила праймер РНК) и ранее синтезированной ДНК, закрываются ферментной ДНК-лигазой, которая катализирует образование фосфодиэфирной связи между 3′-ОН концом одного нуклеотида и 5 ‘Фосфатный конец другого фрагмента.
В таблице приведены ферменты, участвующие в репликации прокариотической ДНК, и функции каждого из них.
Репликация прокариотической ДНК: ферменты и их функции : Ферменты, участвующие в репликации прокариотической ДНК, и их функции обобщены в этой таблице.
Репликация ДНК у эукариот
Репликация ДНК у эукариот происходит в три стадии: инициация, удлинение и завершение, которым помогают несколько ферментов.
Цели обучения
Опишите, как ДНК реплицируется у эукариот
Основные выводы
Ключевые моменты
- Во время инициации белки связываются с ориджином репликации, в то время как геликаза раскручивает спираль ДНК, и в ориджине репликации образуются две репликационные вилки.
- Во время элонгации к последовательности праймера добавляются комплементарные нуклеотиды РНК, которые затем заменяются нуклеотидами ДНК.
- Во время удлинения ведущая нить образуется непрерывно, а отстающая нить состоит из частей, называемых фрагментами Окадзаки.

- Во время терминации праймеры удаляются и заменяются новыми нуклеотидами ДНК, а остов запечатывается ДНК-лигазой.
Ключевые термины
- точка начала репликации : конкретная последовательность в геноме, в которой начинается репликация
- ведущая цепь : матричная цепь двойной спирали ДНК, ориентированная так, что репликационная вилка перемещается вдоль нее в направлении от 3 ‘к 5’
- запаздывающая цепь : цепь двойной спирали ДНК-матрицы, ориентированная так, что репликационная вилка перемещается вдоль нее в направлении от 5 ‘к 3’
Поскольку геномы эукариот довольно сложны, репликация ДНК — очень сложный процесс, в котором задействованы несколько ферментов и других белков.Это происходит в три основных этапа: начало, удлинение и завершение.
Инициирование
Эукариотическая ДНК связана с белками, известными как гистоны, с образованием структур, называемых нуклеосомами.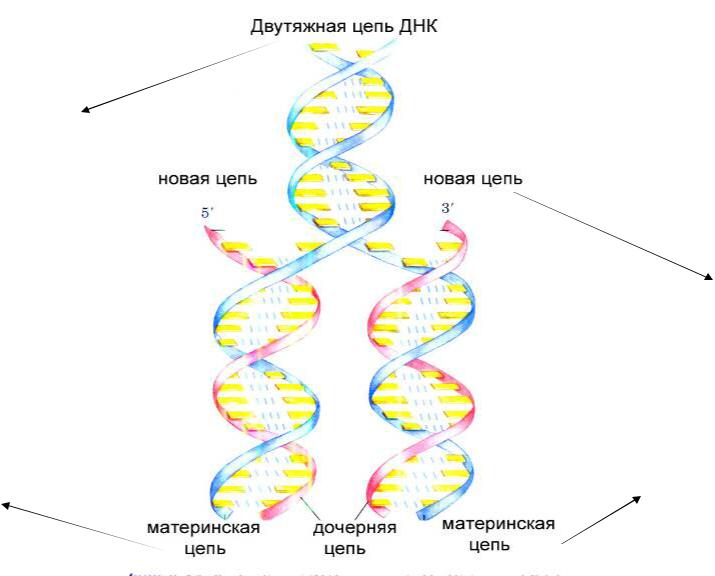 Во время инициации ДНК становится доступной для белков и ферментов, участвующих в процессе репликации. Есть определенные места в хромосоме, называемые источниками репликации, где начинается репликация. У некоторых эукариот, таких как дрожжи, эти местоположения определяются наличием определенной последовательности пар оснований, с которыми связываются белки инициации репликации.У других эукариот, таких как люди, по-видимому, нет согласованной последовательности для происхождения их репликации. Вместо этого белки инициации репликации могут идентифицировать и связываться со специфическими модификациями нуклеосом в исходной области.
Во время инициации ДНК становится доступной для белков и ферментов, участвующих в процессе репликации. Есть определенные места в хромосоме, называемые источниками репликации, где начинается репликация. У некоторых эукариот, таких как дрожжи, эти местоположения определяются наличием определенной последовательности пар оснований, с которыми связываются белки инициации репликации.У других эукариот, таких как люди, по-видимому, нет согласованной последовательности для происхождения их репликации. Вместо этого белки инициации репликации могут идентифицировать и связываться со специфическими модификациями нуклеосом в исходной области.
Некоторые белки распознают и связываются с источником репликации, а затем позволяют другим белкам, необходимым для репликации ДНК, связываться с той же областью. Говорят, что первые белки, связывающиеся с ДНК, «рекрутируют» другие белки. Две копии фермента, называемого геликазой, входят в число белков, привлеченных к источнику.Каждая геликаза раскручивается и разделяет спираль ДНК на одноцепочечную ДНК. Когда ДНК открывается, образуются Y-образные структуры, называемые репликационными вилками. Поскольку две геликазы связываются, в начале репликации образуются две вилки репликации; они расширяются в обоих направлениях по мере того, как репликация продолжается, создавая репликационный пузырь. На хромосоме эукариот существует несколько источников репликации, которые позволяют репликации происходить одновременно в сотнях и тысячах мест вдоль каждой хромосомы.
Когда ДНК открывается, образуются Y-образные структуры, называемые репликационными вилками. Поскольку две геликазы связываются, в начале репликации образуются две вилки репликации; они расширяются в обоих направлениях по мере того, как репликация продолжается, создавая репликационный пузырь. На хромосоме эукариот существует несколько источников репликации, которые позволяют репликации происходить одновременно в сотнях и тысячах мест вдоль каждой хромосомы.
Формирование вилки репликации : вилка репликации образуется открытием точки начала репликации; геликаза разделяет нити ДНК. Праймер РНК синтезируется примазой и удлиняется ДНК-полимеразой. На ведущей цепи необходим только один праймер РНК, и ДНК синтезируется непрерывно, тогда как на отстающей цепи ДНК синтезируется короткими отрезками, каждый из которых должен начинаться со своего собственного праймера РНК. Фрагменты ДНК соединены ДНК-лигазой (не показано).
Удлинение
Во время удлинения фермент, называемый ДНК-полимеразой, добавляет нуклеотиды ДНК к 3′-концу вновь синтезированной полинуклеотидной цепи. Цепь матрицы указывает, какой из четырех нуклеотидов ДНК (A, T, C или G) добавляется в каждое положение новой цепи. К новой цепи добавляется только нуклеотид, комплементарный матричному нуклеотиду в этом положении.
Цепь матрицы указывает, какой из четырех нуклеотидов ДНК (A, T, C или G) добавляется в каждое положение новой цепи. К новой цепи добавляется только нуклеотид, комплементарный матричному нуклеотиду в этом положении.
содержит бороздку, которая позволяет ей связываться с одноцепочечной матричной ДНК и перемещаться по одному нуклеотиду за раз.Например, когда ДНК-полимераза встречает аденозиновый нуклеотид на матричной цепи, она добавляет тимидин к 3′-концу вновь синтезированной цепи, а затем перемещается к следующему нуклеотиду на матричной цепи. Этот процесс будет продолжаться до тех пор, пока ДНК-полимераза не достигнет конца цепи матрицы.
ДНК-полимераза не может инициировать синтез новой цепи; он только добавляет новые нуклеотиды на 3′-конце существующей цепи. Все вновь синтезированные полинуклеотидные цепи должны быть инициированы специализированной РНК-полимеразой, называемой примазой.Примаза инициирует синтез полинуклеотидов и путем создания короткой полинуклеотидной цепи РНК, комплементарной цепи матричной ДНК.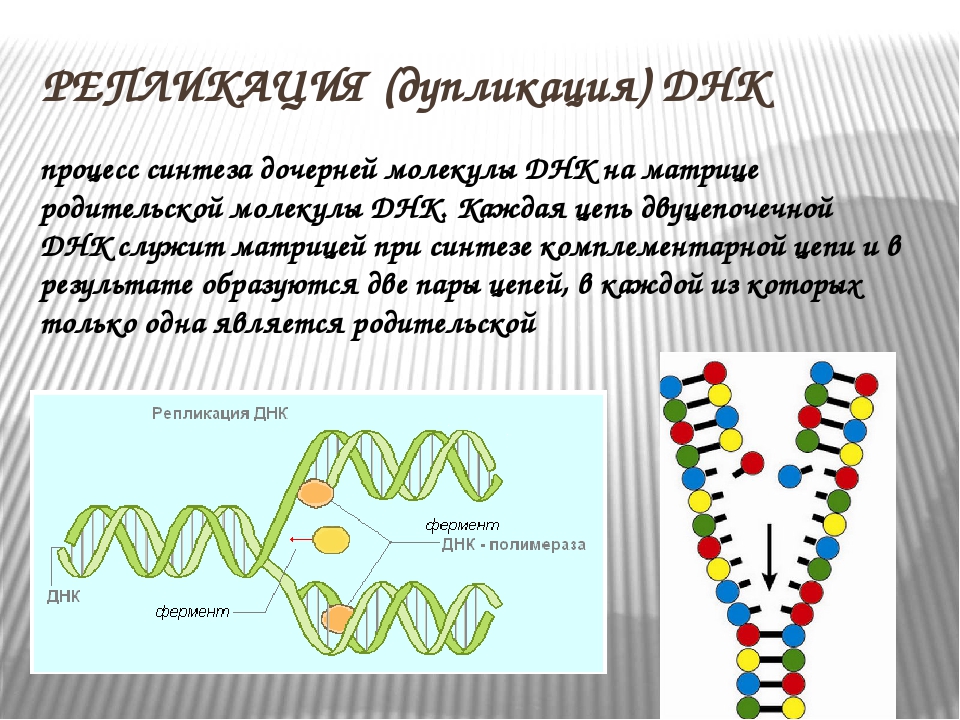 Этот короткий участок нуклеотидов РНК называется праймером. После того, как праймер РНК был синтезирован в матричной ДНК, примаза выходит, и ДНК-полимераза удлиняет новую цепь нуклеотидами, комплементарными матричной ДНК.
Этот короткий участок нуклеотидов РНК называется праймером. После того, как праймер РНК был синтезирован в матричной ДНК, примаза выходит, и ДНК-полимераза удлиняет новую цепь нуклеотидами, комплементарными матричной ДНК.
В конце концов, нуклеотиды РНК в праймере удаляются и заменяются нуклеотидами ДНК. После завершения репликации ДНК дочерние молекулы полностью состоят из непрерывных нуклеотидов ДНК без частей РНК.
Ведущие и отстающие нити
ДНК-полимеразаможет синтезировать новые цепи только в направлении от 5 ‘к 3’. Следовательно, две вновь синтезированные нити растут в противоположных направлениях, поскольку нити-матрицы на каждой репликационной вилке антипараллельны. «Ведущая цепь» непрерывно синтезируется по направлению к репликационной вилке, поскольку геликаза раскручивает матричную двухцепочечную ДНК.
«Отстающая цепь» синтезируется в направлении от вилки репликации и от раскручивающейся ДНК-геликазы.Эта отстающая цепь синтезируется по частям, потому что ДНК-полимераза может синтезировать только в направлении от 5 ‘к 3’, и поэтому она постоянно сталкивается с ранее синтезированной новой цепью. Эти части называются фрагментами Окадзаки, и каждый фрагмент начинается со своего собственного праймера РНК.
Эти части называются фрагментами Окадзаки, и каждый фрагмент начинается со своего собственного праймера РНК.
Прекращение
Хромосомы эукариот имеют несколько источников репликации, которые инициируют репликацию почти одновременно. Каждая точка начала репликации образует пузырь из дублированной ДНК по обе стороны от точки начала репликации.В конце концов, ведущая нить одного репликационного пузыря достигает отстающей нити другого пузыря, а отстающая нить достигает 5′-конца предыдущего фрагмента Окадзаки в том же пузыре.
ДНК-полимераза останавливается, когда достигает участка матрицы ДНК, который уже был реплицирован. Однако ДНК-полимераза не может катализировать образование фосфодиэфирной связи между двумя сегментами новой цепи ДНК, и она разрывается. Эти незакрепленные участки сахарно-фосфатного остова в полностью реплицированной цепи ДНК называются зарубками.
После того, как все нуклеотиды матрицы реплицированы, процесс репликации еще не завершен. Праймеры РНК необходимо заменить ДНК и соединить разрывы в сахарно-фосфатном скелете.
Праймеры РНК необходимо заменить ДНК и соединить разрывы в сахарно-фосфатном скелете.
Группа клеточных ферментов, удаляющих праймеры РНК, включает белки FEN1 (эндонульцеаза лоскута 1) и РНКаза Н. Ферменты FEN1 и РНКаза Н удаляют праймеры РНК в начале каждой ведущей цепи и в начале каждого фрагмента Окадзаки, оставляя пробелы. нереплицированной матричной ДНК.Как только праймеры удалены, свободно плавающая ДНК-полимераза приземляется на 3′-конец предыдущего фрагмента ДНК и удлиняет ДНК через промежуток. Однако это создает новые зазубрины (несвязанный сахарно-фосфатный каркас).
На заключительной стадии репликации ДНК энзимлигаза присоединяется к сахарно-фосфатным остовам в каждом месте разрыва. После того, как лигаза соединила все разрывы, новая цепь представляет собой одну длинную непрерывную цепь ДНК, а дочерняя молекула ДНК завершена.
Репликация ДНК : Это отрывок из производства PBS под названием «ДНК: Секрет жизни. В нем подробно рассказывается о последних исследованиях (по состоянию на 2005 год), касающихся процесса репликации ДНК.
В нем подробно рассказывается о последних исследованиях (по состоянию на 2005 год), касающихся процесса репликации ДНК.
Репликация теломер
Поскольку ДНК-полимераза сама по себе не может реплицировать концы хромосом, теломераза помогает в их репликации и предотвращает деградацию хромосом.
Цели обучения
Опишите роль теломеразы в репликации теломер
Основные выводы
Ключевые моменты
- ДНК-полимераза не может реплицировать и восстанавливать молекулы ДНК на концах линейных хромосом.
- Концы линейных хромосом, называемые теломерами, защищают гены от удаления при продолжении деления клеток.
- Фермент теломераза прикрепляется к концу хромосомы; комплементарные основания к матрице РНК добавляются на 3′-конце цепи ДНК.
- Как только отстающая цепь удлиняется теломеразой, ДНК-полимераза может добавлять комплементарные нуклеотиды к концам хромосом, и теломеры, наконец, могут реплицироваться.

- Теломеры клеток, которые претерпевают деление, продолжают укорачиваться, поскольку большинство соматических клеток не вырабатывают теломеразу; Укорочение теломер связано со старением.
- Реактивация теломеразы у мышей с дефицитом теломеразы вызывает удлинение теломер; это может иметь потенциал для лечения возрастных заболеваний у людей.
Ключевые термины
- теломер : любая из повторяющихся нуклеотидных последовательностей на каждом конце эукариотической хромосомы, которые защищают хромосому от деградации
- теломераза : фермент в эукариотических клетках, который добавляет определенную последовательность ДНК к теломерам хромосом после их деления, обеспечивая стабильность хромосом во времени
Конец проблемы линейной репликации ДНК
Линейные хромосомы имеют конечную проблему.После репликации ДНК каждая вновь синтезированная цепь ДНК на своем 5′-конце короче, чем на 5′-конце родительской цепи ДНК. Это создает 3′-выступ на одном конце (и только на одном конце) каждой дочерней цепи ДНК, так что две дочерние ДНК имеют свои 3′-выступы на противоположных концах
Это создает 3′-выступ на одном конце (и только на одном конце) каждой дочерней цепи ДНК, так что две дочерние ДНК имеют свои 3′-выступы на противоположных концах
Проблема конца теломер : Упрощенная схема репликации ДНК, где родительская ДНК (вверху) реплицируется из трех источников репликации, давая три репликационных пузыря (в центре) перед тем, как дать начало двум дочерним ДНК (внизу).Нити родительской ДНК — черные, вновь синтезированные цепи ДНК — синие, а праймеры РНК — красные. Все праймеры РНК удаляются РНКазой H и FEN1, оставляя промежутки во вновь синтезированных цепях ДНК (не показаны). ДНК-полимераза и лигаза заменят все праймеры РНК ДНК, за исключением праймера РНК на 5′-концах каждой новой -синтезированная (синяя) прядь. Это означает, что каждая вновь синтезированная цепь ДНК на своем 5′-конце короче, чем эквивалентная цепь в родительской ДНК.
Каждый праймер РНК, синтезируемый во время репликации, может быть удален и заменен цепями ДНК, за исключением праймера РНК на 5′-конце вновь синтезированной цепи. Этот небольшой участок РНК можно только удалить, но не заменить ДНК. Ферменты РНКаза H и FEN1 удаляют праймеры РНК, но ДНК-полимераза добавит новую ДНК только в том случае, если у ДНК-полимеразы есть существующая цепь 5 ‘к ней («позади») для удлинения. Однако в 5′-направлении после конечного праймера РНК больше нет ДНК, поэтому ДНК-полимера не может заменить РНК ДНК. Следовательно, обе дочерние цепи ДНК имеют неполную 5′-цепь с 3’-выступом.
Этот небольшой участок РНК можно только удалить, но не заменить ДНК. Ферменты РНКаза H и FEN1 удаляют праймеры РНК, но ДНК-полимераза добавит новую ДНК только в том случае, если у ДНК-полимеразы есть существующая цепь 5 ‘к ней («позади») для удлинения. Однако в 5′-направлении после конечного праймера РНК больше нет ДНК, поэтому ДНК-полимера не может заменить РНК ДНК. Следовательно, обе дочерние цепи ДНК имеют неполную 5′-цепь с 3’-выступом.
В отсутствие дополнительных клеточных процессов нуклеазы переваривали бы эти одноцепочечные 3′-выступы.Каждая дочерняя ДНК станет короче родительской, и в конечном итоге вся ДНК будет потеряна. Чтобы предотвратить это укорочение, концы линейных эукариотических хромосом имеют особые структуры, называемые теломерами.
Репликация теломер
Концы линейных хромосом известны как теломеры: повторяющиеся последовательности, которые не кодируют конкретный ген. Эти теломеры защищают важные гены от делетирования при делении клеток и укорочении цепей ДНК во время репликации.
У человека последовательность из шести пар оснований TTAGGG повторяется от 100 до 1000 раз. После каждого раунда репликации ДНК некоторые теломерные последовательности теряются на 5′-конце вновь синтезированной цепи каждой дочерней ДНК, но поскольку это некодирующие последовательности, их потеря не оказывает отрицательного воздействия на клетку. Однако даже эти последовательности не безграничны. После достаточного количества циклов репликации все теломерные повторы теряются, и ДНК рискует потерять кодирующие последовательности с последующими циклами.
Открытие фермента теломеразы помогло понять, как поддерживаются концы хромосом. Фермент теломераза прикрепляется к концу хромосомы и содержит каталитическую часть и встроенную матрицу РНК. Теломераза добавляет основания комплементарной РНК к 3′-концу цепи ДНК. Как только 3′-конец матрицы отстающей цепи становится достаточно удлиненным, ДНК-полимераза добавляет комплементарные нуклеотиды к концам хромосом; таким образом, концы хромосом реплицируются.
Теломераза важна для поддержания целостности хромосом. : Концы линейных хромосом поддерживаются действием фермента теломеразы.
Теломераза и старение
Теломераза обычно активна в половых клетках и взрослых стволовых клетках, но не активна в соматических клетках взрослых. В результате теломераза не защищает ДНК взрослых соматических клеток, и их теломеры постоянно укорачиваются по мере того, как они подвергаются циклам клеточного деления.
В 2010 году ученые обнаружили, что теломераза может изменить некоторые возрастные заболевания у мышей.Эти открытия могут внести свой вклад в будущее регенеративной медицины. В исследованиях ученые использовали мышей с дефицитом теломеразы с атрофией тканей, истощением стволовых клеток, органной недостаточностью и нарушением реакции на повреждение тканей. Реактивация теломеразы у этих мышей вызвала удлинение теломер, уменьшила повреждение ДНК, обратила вспять нейродегенерацию и улучшила функцию яичек, селезенки и кишечника.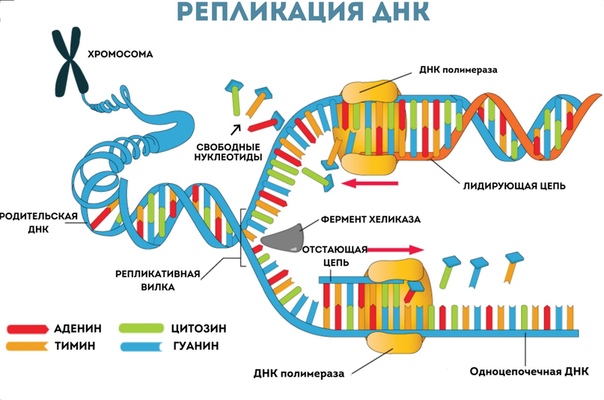 Таким образом, реактивация теломер может иметь потенциал для лечения возрастных заболеваний у людей.
Таким образом, реактивация теломер может иметь потенциал для лечения возрастных заболеваний у людей.
Нанотехнологии и двойная спираль
В 2003 году исполнилось 50 лет со дня открытия Джеймсом Д. Уотсоном и Фрэнсисом Х. Криком структуры двойной спирали ДНК. Их открытие свело генетику к химии и заложило основы биологии на следующие полвека. Сегодня тысячи исследователей усердно работают над разгадыванием бесчисленных способов, которыми гены контролируют развитие и функционирование организмов. Все эти гены записаны в среде, которой является ДНК.
Однако у этой необычной молекулы есть и другие применения помимо биохимии. Используя методы современной биотехнологии, мы можем создавать длинные молекулы ДНК с последовательностью строительных блоков, выбранных по желанию. Эта способность открывает дверь к новым путям, на которые не пошла природа при развитии жизни. В 1994 году, например, Леонард М. Адлеман из Университета Южной Калифорнии продемонстрировал, как ДНК можно использовать в качестве вычислительного устройства. В этой статье я расскажу о другом небиологическом использовании ДНК: построении структур и устройств, основные элементы и механизмы которых имеют размер от одного до 100 нанометров — одним словом, нанотехнологии.
Такие конструкции имеют множество потенциальных применений. Регулярные решетки из ДНК могут удерживать копии больших биологических молекул в упорядоченном массиве для рентгеновской кристаллографии для определения их структуры, что является важным шагом в рациональном дизайне лекарств. В качестве альтернативы решетки могут служить каркасом для наноэлектронных компонентов, либо в качестве рабочего устройства, либо в качестве шага в производстве устройства. Материалы могут быть созданы — либо из ДНК, либо из нее — со структурами, точно спроектированными на молекулярном уровне.Машины ДНК с движущимися частями могут использоваться в качестве наномеханических датчиков, переключателей и пинцетов, а также для более сложных роботизированных функций.
Разветвленная ДНК
НАНОМАСШТАБ — это шкала молекул. Типичная связь между двумя атомами составляет около 0,15 нанометра. (Нанометр равен одной миллиардной метра.) Спираль ДНК имеет диаметр около двух нанометров, и она делает полный круг каждые 3,5 нанометра или около того, на расстоянии около 10 пар оснований, которые образуют ступеньки лестницы ДНК. [ см. Рамку вверху на странице 33 ].Короткий фрагмент ДНК имеет высокоспецифические взаимодействия с другими химическими веществами в зависимости от последовательности пар оснований. Можно представить себе использование таких частей для распознавания определенных молекул или для управления составом материала, действуя как катализатор. И в течение многих лет биологи использовали ДНК для ее распознавания, особенно используя липкие концы в генной инженерии. Липкий конец возникает, когда одна нить спирали выходит на несколько непарных оснований за пределы другой [ см. Иллюстрацию внизу на противоположной странице ].Липкость — это склонность нависающей части соединяться с соответствующей цепью, которая имеет дополнительные основания в соответствующем порядке — аденин на одной цепи соединяется с тимином на противоположной цепи, а цитозин связывается с гуанином.
На первый взгляд кажется, что ДНК не может приводить к интересным структурам. Природная ДНК образует линейную цепочку, похожую на длинный кусок бечевки, так что все, что можно себе представить, — это линии или круги, возможно, так или иначе скрученные или завязанные узлами.Но линейная цепь — не единственная форма, которую принимает ДНК. Во время определенных клеточных процессов ДНК кратковременно существует в виде разветвленной молекулы. Это ветвление происходит при репликации ДНК (при подготовке к делению клетки) и во время рекомбинации (когда генетический материал меняется между совпадающими парами хромосом, как это происходит при производстве спермы и яйцеклеток).
Ветви образуются, когда двойная спираль частично распадается на две нити. При репликации каждая цепь превращается в новую двойную спираль путем добавления комплементарных нуклеотидов по всей ее длине.(Нуклеотид — это комбинация основания и соответствующего участка скелета спирали.) Более интересным является кроссовер, который происходит при рекомбинации, при которой два фрагмента ДНК разрываются и частично распадаются, и в результате четыре нити соединяются примерно так же, как перекресток на пересечении двух автомагистралей.
При рекомбинации ДНК возникает точка ветвления, когда каждая из четырех цепей переключается с одного партнера на другого. Точка ветвления перемещается из-за двойной симметрии (как у цифры 69) в базовых последовательностях, которые ее фланкируют.Эта симметрия означает, что каждая прядь может сочетаться с любой из двух других прядей. В 1979 году я работал с Брюсом Х. Робинсоном, который сейчас работает в Вашингтонском университете, чтобы описать природу этого движения, когда я понял, что синтетические молекулы ДНК, лишенные этой симметрии, могут образовывать разветвленные молекулы, точки ветвления которых не перемещаются. Чтобы спроектировать такое соединение, нужно сделать четыре нити ДНК. Для каждой нити последовательность вдоль половины нити будет соответствовать половине второй нити, а оставшаяся половина будет соответствовать половине третьей нити.
Излюбленная структураДНК — обычная двойная спираль, идентифицированная Уотсоном и Криком. Величина, называемая свободной энергией, определяет, какая структура предпочтительна. В общем, свободная энергия определяет, идет ли химическая реакция в прямом или обратном направлении; он также определяет конформацию — складки и соединения — больших молекул, таких как ДНК, РНК и белки. Химическая система всегда стремится перейти в состояние с наименьшей свободной энергией. Для двух комплементарных цепей нуклеотидов свободная энергия сводится к минимуму, когда они соединяются в пару с образованием двойной спирали.
Четыре нити нашего неподвижного соединения могут соединиться и образовать максимальное количество обычных двойных спиралей ДНК, только образуя разветвленную молекулу. В общем, точка ветвления не приветствуется — она увеличивает свободную энергию молекулы — но это увеличение перевешивается гораздо большей экономией энергии в четырех плечах, сделанных из обычной двойной спирали ДНК. Сегодня синтезировать такие цепи и реализовать идею стабильной разветвленной молекулы ДНК просто, но в 1979 году это была современная химия, и я был кристаллографом, а не химиком-органиком, поэтому в основном я просто думал о система.(Только в 1982 году я узнал, как создавать ДНК.)
Вдохновение от Эшера
Я СОГЛАСИЛ, что должно быть возможно создавать разветвленные соединения ДНК с множеством плеч, а не только с четырьмя. Однажды, осенью 1980 года, я зашел в паб на кампусе, чтобы подумать о «перекрестках с шестью рукавами». Почему-то я подумал о голландском художнике М. К. Эшерсе, гравюра на дереве Глубина [ см. Иллюстрацию на странице 34 ]. Я понял, что центр каждой рыбы на этой картинке был как идеализированное изображение точки ветвления шестиконечного соединения.Шесть элементов выходят из этой центральной точки на рыбе: голова и хвост, верхний и нижний плавники, левый и правый плавники. Рыбы устроены так же, как молекулы в молекулярном кристалле, с регулярными повторениями вперед и назад, вверх и вниз, влево и вправо. Меня поразило, что, если я скреплю стыки липкими концами, я смогу организовать материю в нанометровом масштабе так же, как Эшер скреплял косяк рыб, используя свое воображение.
У нас есть несколько веских причин для строительства таких сооружений.Во-первых, мы стремимся изготавливать макроскопические частицы материи, состоящие из спроектированных молекул, объединенных вместе в структуру, управляемую с наноскопической точностью. Эта процедура может привести к получению материалов с новыми свойствами или новыми комбинациями свойств. Например, материалы с заданными оптическими свойствами, такие как фотонные кристаллы, могут быть изготовлены путем создания точно определенных массивов с конкретными повторяющимися расстояниями.
Другая цель — использовать ДНК в качестве каркаса для удержания других молекул в массивах, включая те, которые сами по себе не образуют регулярную кристаллическую структуру.Таким образом, можно было изготавливать кристаллы для использования в экспериментах по кристаллографии, создавая клетки ДНК, содержащие внутри себя большие биологические молекулы, такие как белки [ см. Правую иллюстрацию на следующей странице ]. Такие клетки позволят кристаллографам определять трехмерные структуры заключенных в них молекул — ключевую процедуру в рациональном дизайне лекарств, которые точно взаимодействуют с определенными частями целевой молекулы. (Это кристаллографическое приложение наиболее сильно мотивирует мой интерес к этой области.В настоящее время многие рецепторные молекулы, которые могут быть отличными мишенями для лекарств, не поддаются традиционной кристаллографии. Подобным образом можно организовать наноэлектронные компоненты в очень маленькие устройства памяти, как мы с Робинсоном предложили в 1987 году.
Зачем использовать ДНК для этих целей? Основная причина в том, что нити ДНК взаимодействуют наиболее программируемым и предсказуемым образом. Липкий конец, который имеет длину N и оснований, имеет одну из 4 N возможных последовательностей оснований.Эта огромная изменчивость и склонность конца связываться только с близко соответствующей последовательностью предоставляют широкие возможности для создания молекул, которые состоят из большого количества цепей ДНК, соединенных друг с другом полностью определенным образом. Кроме того, мы знаем, что два липких конца образуют классическую спиральную структуру ДНК, когда они сцепляются, и эти спиральные участки ДНК относительно жесткие. Таким образом, мы знаем не только, какие пряди связаны с какими другими прядями, но и детальную форму соединенных сегментов.С середины 1990-х годов стало возможным программировать формы разветвленных видов ДНК, используя только их последовательности. У нас нет такой конкретной информации о белках или антителах, которые являются другими кандидатами на рабочие элементы. Они также имеют огромную вариативность, но определение того, какую форму примет белок и как два белка или антитела будут соединяться вместе, является трудоемкой задачей, которую придется решать заново для каждого примера.
Еще одна причина для работы с ДНК — простота ее синтеза с помощью инструментов биотехнологической индустрии.Мы можем манипулировать ДНК с помощью множества ферментов, таких как рестрикционные ферменты (которые расщепляют ДНК на определенных участках) или лигазы (которые катализируют соединение двух молекул ковалентными связями — прочными химическими связями, которые включают обмен парами электронов между атомами). Эти инструменты можно использовать для создания и манипулирования традиционной ДНК, а также экзотических производных, в которые включены различные основания из четырех обычных или в которые дополнительные молекулы прикреплены за пределами остова ДНК (стороны лестницы ДНК) .Медицинские исследователи, надеющиеся использовать нуклеиновые кислоты (ДНК и РНК) для терапии, сделали множество таких вариантов. ДНК чрезвычайно хорошо подходит для создания таких производных, потому что каждый нуклеотид вдоль спирали имеет участки, к которым могут быть прикреплены молекулы.
Наконец, как мы увидим ниже, ДНК можно заставить образовывать структуры, отличные от стандартной двойной спирали. Мы можем создавать наномеханические устройства, части которых движутся — например, закрывающий пинцет или вращающийся вал — при переходе от одной структуры ДНК к другой.Одним из недостатков является то, что объекты ДНК должны быть сконструированы в водном растворе. Однако высушить полученные структуры (например, на слюде) не проблема, как мы делаем это для получения микроскопических изображений наших результатов.
Stick Models
ПЕРВЫМ ШАГОМ в любой новой программе научных исследований является определение базовой осуществимости проекта. В 1991 году мы с Чжунхуэем Ченом из Университета Делавэра сделали это, построив молекулу ДНК в форме куба, сформированного из палочек [ см. Иллюстрацию ниже ].Каждый край куба представляет собой отрезок двойной спирали ДНК; каждый угол представляет собой соединение с тремя рукавами. Каждый угол соединяется с тремя другими углами; Говорят, что подключений кубиков три. Генные инженеры создали множество линейных конструкций ДНК, но это была первая молекула ДНК с связностью более двух. Куб самостоятельно собирается из кусочков ДНК, предназначенных для сцепления друг с другом, но концы каждого фрагмента не соединяются. Лигазы могут соединять эти свободные концы, в результате чего получается шесть замкнутых петель, по одной для каждой грани куба.Из-за спиральной природы ДНК каждая из этих петель закручена вокруг петель, которые ее окружают, поэтому куб не может развалиться, даже если все связи, соединяющие пары оснований, каким-то образом были разорваны.
Ювэнь Чжан, сейчас из Human Genome Sciences в Роквилле, штат Мэриленд, и я построили еще одну форму, названную усеченным октаэдром, которая похожа на куб, но более сложна [ см. Иллюстрацию на странице 30 ]. Хотя трехлепестковых соединений было бы достаточно для создания отдельных усеченных октаэдров, вместо этого мы построили их, используя четырехлепестковые соединения.Мы предполагали, что дополнительное плечо, выступающее в каждом углу, может быть использовано для соединения усеченных октаэдров вместе в более крупную структуру, но в конце концов мы не пошли в этом направлении. Мы создали очень крошечное количество усеченных октаэдров — достаточно, чтобы охарактеризовать их структуру, но слишком мало, чтобы попытаться соединить их вместе — и даже этот крошечный образец привел нас к пределам того, что мы могли сделать без капитального ремонта наших процедур ( например, путем роботизации повторяющихся шагов). Вместо этого мы обратились к более простым компонентам.
Другой причиной изменения направления было то, что по пути мы поняли, что построенные нами многогранники не жесткие. ДНК — это жесткая молекула: отрезок ДНК длиной в два или три витка (длины, которые мы используем для краев многогранников) может покачиваться вокруг оси своей спирали не более, чем кусок приготовленных спагетти длиной два или три миллиметра может покачиваться вокруг своей оси. центральная ось. Эта негибкость гарантировала, что края наших фигурок были жесткими, но мы узнали, что углы в каждом углу были довольно разными.Построенные нами многогранники были похожи на конструкции из зубочисток, вбитых в шарики зефира по углам. Такие конструкции могут иметь применение, но построение регулярной решетки не входит в их число. Гораздо проще самостоятельно собрать упорядоченный кристаллический кусок материи из кирпичиков, чем из зефира.
Чтобы решить эту проблему, моя группа исследовала другой разветвленный мотив, обнаруженный в системах биологической рекомбинации, молекулу двойного кроссовера ДНК (DX). Молекула DX состоит из двух двойных спиралей, выровненных бок о бок, с нитями, пересекающимися между спиралями, связывающими их вместе [ см. Рамку на следующей странице ].Мы охарактеризовали эту молекулу и установили, что она жесткая. Мы также продемонстрировали, что молекула DX, содержащая еще одну небольшую двойную спиральную область (называемая молекулой DX + J), очень жесткая. Эта дополнительная двойная спиральная область создает выпуклость на вершине молекулы DX, которая служит маркером — нанотехнологическим эквивалентом капли краски.
В сотрудничестве с Эриком Винфри из Калифорнийского технологического института, Фуронг Лю и Лиза А. Венцлер из моей группы в Нью-Йоркском университете использовали комбинации молекул DX и DX + J в качестве плиток для создания двумерных кристаллов с заданными узорами.Плитки соединены липкими концами на каждой спирали. Одна компоновка, при которой столбцы плиток DX чередуются со столбцами плиток DX + J, дает узор из полос, разделенных примерно 32 нанометрами. Мы разместили массивы на плоской поверхности слюды и исследовали их с помощью атомно-силового микроскопа, чтобы подтвердить, что структура имеет правильные размеры. Мы установили, что узор не был случайным, создав второй кристалл с модифицированными плитками, которые соединяются вместе с тремя столбцами DX для каждого столбца DX + J, чтобы получить полосы с двойным разделением.
Совсем недавно группа Джона Х. Рейфа из Университета Дьюка продемонстрировала штрих-коды ДНК, сделанные с использованием таких рисунков. В этих мозаиках положения полос были запрограммированы так, чтобы они появлялись в шаблоне, представляющем число 01101 (с молекулами, аналогичными нашим DX и DX + J, которые служат как 0 и 1, соответственно). Паттерн был запрограммирован с использованием входной цепи ДНК, последовательность которой кодировала паттерн 01101. Аналоги кирпичей DX и DX + J самоорганизовывались на участках цепи ДНК, соответствующих 0 и 1 соответственно.Многие такие последовательности из пяти кирпичей затем соединились параллельно, образуя узор из полос 01101. Расстояние между полосами составляло примерно 15 нанометров. Изучая полоски с помощью атомно-силового микроскопа, можно эффективно использовать штрих-код для считывания данных, которые были закодированы на входной цепи ДНК. Это визуальное средство считывания последовательности ДНК могло бы значительно ускорить этап считывания вычислений на основе ДНК, а также могло бы использоваться для картирования мутаций. В недавнем захватывающем расширении использования длинных цепей ДНК Пол Ротемунд из Калифорнийского технологического института использовал вирусную цепочку из примерно 7000 нуклеотидов для построения сложных паттернов, включая смайлик и карту Западного полушария.
Чэндэ Мао, сейчас работающий в Университете Пердью, и я сделали из параллелограммов ДНК двухмерные узоры, похожие на наши многогранники из палочек. Копии этого устройства могут быть соединены в кристалл, который простирается как вафля в двух измерениях. Можно настроить размеры полостей в массиве, изменив размеры параллелограммов. Хотя отдельные разветвленные соединения являются гибкими, расположение четырех из них по углам параллелограмма приводит к созданию хорошо функционирующего элемента в массиве параллелограммов.
Наномашины
ЦЕНТРАЛЬНЫЕ НАНОТЕХНОЛОГИИ — это машины молекулярного масштаба. ДНК оказалась очень полезной для создания этих машин. Мы создали несколько устройств из ДНК, но здесь я остановлюсь на двух, которые имеют четко определенные структуры. В обоих случаях механизм основан на структурном переходе молекул ДНК — переходе от одной конформации (такой как обычная двойная спираль) к другой.
Обычная ДНК представляет собой правую спираль. Представьте, что вы поднимаетесь по винтовой лестнице, держась левой рукой за внутренние перила, а правую — за внешние.Такая лестница представляет собой правую спираль. Обычная правая ДНК называется B-ДНК и является наиболее энергетически предпочтительной структурой в типичных водных условиях.
Двухспиральная ДНК может также иметь множество различных структур в зависимости от последовательности ее оснований и химических соединений, присутствующих в растворе, в который она погружена. Одним из них является Z-ДНК, структура которой была впервые описана в 1979 году Александром Ричем и его коллегами из Массачусетского технологического института [ см. Рамку вверху на странице 33 ].Z-ДНК представляет собой левостороннюю структуру ДНК.
Для создания Z-ДНК обычно требуется участок чередующихся оснований цитозина и гуанина. Каркас ДНК включает отрицательно заряженные фосфатные группы, которые в структуре Z-ДНК сближаются. Такое образование благоприятно только в том случае, если заряды фосфатов могут быть экранированы друг от друга водной средой, содержащей либо высокую концентрацию соли, либо особые эффекторные частицы, такие как гексаммин кобальта, Со (NH 3 ) 6 + ++, который выполняет ту же работу при гораздо меньшей концентрации.Требование последовательности цитозин-гуанин позволяет нам контролировать , где на молекуле ДНК имеет место переход BZ (и, следовательно, то, что делает наша машина), а требования окружающей среды позволяют нам контролировать , когда переход (и, следовательно, действие машины ) происходит.
Мой N.Y.U. коллеги Вэйцюн Сунь и Чжийонг Шэнь, Мао и я построили устройство, состоящее из двух молекул DX, соединенных стержнем из двойной спирали ДНК [ см. иллюстрацию справа ].В середине вала находится последовательность из 20 пар, которые могут принять Z-образную структуру в соответствующих условиях. В обычных условиях каждая часть устройства будет формировать B-ДНК, и две молекулы DX будут находиться с одной стороны от оси валов. Когда в раствор добавляется гексаммин кобальта, центральная часть стержня превращается в Z-ДНК, и одна молекула DX вращается примерно на 3,5 оборота относительно другой; нечетный пол-оборота означает, что теперь они находятся по разные стороны от оси валов. Удаление гексаммина кобальта возвращает устройству его первоначальную структуру.Мы продемонстрировали, что движение происходит с помощью спектроскопии с участием двух цветных красителей, прикрепленных к молекулам DX.
Это устройство B-Z довольно надежно, но имеет недостаток. Если бы группа различных устройств B-Z была включена в большую надстройку (например, одну из двухмерных решеток, обсуждаемых ранее), вся структура имела бы только два состояния: каждая машина в состоянии B или каждая из них в состоянии Z. Для индивидуального управления набором машин требуются устройства с независимыми триггерами.С ДНК, конечно, есть естественный способ сделать это, используя нити ДНК в качестве триггеров и имея разные последовательности оснований для запуска каждой машины.
Чтобы реализовать эту схему, Хао Янь из Университета штата Аризона, Сяопин Чжан из Нью-Йорка, Шэнь и я разработали систему, которая меняет форму, когда с ней связываются разные нити. Система состоит из двух параллельных двойных спиралей ДНК, каждая из которых сводится к одной цепи в центральной области кроссовера. Кроссовер участок может принимать два разных состояния, в соответствии с которыми конкретные цепи были добавлены к раствору для связывания с одноцепочечными участками [ см. Рамку на следующей странице ].Два состояния устройства называются PX (паранемический кроссовер) и JX (совмещены). Когда устройство находится в состоянии PX, две спирали на одной стороне центрального соединения поворачиваются примерно на пол-оборота от их положения в состоянии JX.
Добавление к раствору определенной пары нитей (называемых набором нитей) переводит устройство в состояние JX, связываясь с центральной областью без кроссинговера. Чтобы перейти в состояние PX, мы должны сначала удалить эти установленные пряди. В 2000 году Бернард Юрке и его коллеги из Lucent Technologies показали, что цепь может быть извлечена из ДНК путем связывания цепей в полном объеме с ней.Чтобы реализовать этот процесс, у наших наборов прядей есть короткие концы, которые не соединяются с машиной. Когда мы добавляем в раствор полную дополнительную нить, она начинается с присоединения к непарному удлинению, а затем отделяется от оставшейся части установленной нити с устройства.
Удалив первые прядки из рамы, мы можем добавить другие прядки, которые связываются с центральной областью и пересекаются там. Эта привязка поворачивает две двойные спирали и переводит устройство в состояние PX.Процесс можно обратить, удалив вторую прядь и добавив обратно первые. Таким образом, двойные спирали можно поворачивать вперед и назад по желанию. Ряд различных устройств PX-JX может работать независимо, добавляя и удаляя набор цепей, предназначенных для их индивидуальных областей связывания.
Мы использовали атомно-силовую микроскопию, чтобы проверить, как двигалось наше устройство. Мы сделали длинную цепочку из этих устройств и соединили большой блок ДНК в форме трапеции с одной стороны каждого устройства.Когда все устройства находятся в состоянии PX, трапеции лежат на одной стороне цепи. Когда все находятся в состоянии JX, трапеции меняют стороны зигзагообразно.
В 2000 году Юрке и его коллеги продемонстрировали наноскопический пинцет, сделанный из трех нитей ДНК. Установленные пряди, которые Юрке называет топливными, открывали и закрывали пинцетом. Другие исследователи использовали аналогичные методы для включения активности рибозимов — ферментов, состоящих из РНК. В 1998 году Майкл П. Робинсон и Эндрю Д.Эллингтон из Техасского университета в Остине продемонстрировал 10 000-кратное увеличение активности рибозимов путем добавления соответствующей закрепленной цепи, которая связывалась с рибозимом, изменяя его конформацию.
Ключевой целью было включение устройств ДНК в каркасные массивы. Это первый шаг к наноробототехнике на основе ДНК, включающей сложные движения и разнообразие состояний. Вместе с Баокван Дин, который сейчас работает в Молекулярной литейной Национальной лаборатории Лоуренса Беркли, я сообщил о достижении этой ключевой цели в конце 2006 года.И вместе с Шипингом Ляо, который сейчас работает в Barr Pharmaceuticals, я сообщил о системе с несколькими состояниями, использующей устройство PX-JX, которое преобразует сигналы ДНК в инструкции по сборке полимера. Использование устройств, подобных описанным здесь, позволит нам собирать новые материалы с высокой точностью. Лей Чжу, который сейчас работает в Университете штата Флорида, Джеймс У. Кэнэри и Филип С. Люкман из Нью-Йорка, и я недавно собрали прототип, сделанный из небольшого куска нейлона на основе нуклеиновой кислоты. Когда-нибудь мы надеемся, что сможем создавать новые полимеры и комбинации полимеров с определенной топологией (обмотки их основных цепей) и свойствами.
Будущее
ОСНОВНАЯ ЦЕЛЬ нанотехнологий, основанных на ДНК, состоит в том, чтобы распространить успехи в двух измерениях на три измерения. Когда это будет выполнено, мы продемонстрируем способность конструировать твердые материалы, задав серию последовательностей ДНК и затем комбинируя их. Если системы сильно упорядочены, то кристаллографические эксперименты с молекулами, удерживаемыми в регулярно повторяющейся структуре, упомянутой ранее, будут возможны.
Достижение этой цели в первую очередь предполагает использование ДНК в качестве программируемого компонента, но ни кристаллография, ни наноэлектроника не могут полагаться только на ДНК.Например, наноэлектронные компоненты, такие как металлические наночастицы или углеродные нанотрубки, должны быть объединены с молекулами ДНК в системах и жидких растворах, которые совместимы как с ДНК, так и с другими компонентами. Учитывая разнообразную химическую природу этих молекул, было нелегко организовать металлические наночастицы в массивы ДНК. Моя группа и группа Ричарда А. Киля из Университета Миннесоты и Хао Яна, однако, достигли успеха в этом усилии. Кроме того, даже если наноэлектроника может быть построена путем самосборки ДНК, наномашины в конечном итоге должны взаимодействовать с макроскопическим миром более сложным образом, чем добавление и удаление установленных цепей из раствора.Эта проблема, вероятно, будет сложной.
Нанотехнологическая машина мечты — это машина, которая может копировать. Однако, в отличие от линейной ДНК, разветвленная ДНК не поддается саморепликации. Тем не менее в конце 2003 года Уильям М. Ши, Джоэл Д. Киспе и Джеральд Ф. Джойс из Исследовательского института Скриппса в Ла-Хойя, Калифорния, сделали первый захватывающий шаг к самовоспроизводящимся объектам ДНК. Они построили октаэдр из одной длинной цепи ДНК (около 1700 оснований), используя пять коротких вспомогательных цепей для завершения сборки [ см. Иллюстрацию ниже ].Каждый край октаэдра состоит из двух связанных двойных спиралей ДНК — серии молекул DX и PX. Края были длиной около 14 нанометров каждая, или около четырех витков двойной спирали. Сложенный октаэдр не может воспроизводиться, но в развернутом состоянии длинная цепь легко клонируется миллионы раз с помощью стандартного биотехнологического процесса, называемого ПЦР (полимеразная цепная реакция). Это все еще далеко от репликации, достигаемой каждым живым организмом, но к столетию Уотсона-Крика у нас уже должны быть машины на основе ДНК, которые тоже будут работать.
АВТОР
Надриан К. (Нед) Симан обучался кристаллографии, но его разочарование экспериментом по кристаллизации макромолекул привело его к мысли, что соединения ДНК могут быть использованы в новом подходе к кристаллизации. С тех пор он пытается реализовать эту концепцию и ее побочные продукты. Последние 19 лет Симан работал на химическом факультете Нью-Йоркского университета. Когда в середине 1980-х ему сказали, что он занимается нанотехнологиями, его ответ был похож на ответ М.Журден, главный герой «Молира Буржуа Джентильхомм », обрадовался, обнаружив, что всю свою жизнь говорил прозой.
Репликация ДНК — Структура — Этапы репликации
Репликация ДНК, также известная как se ми-консервативная репликация , представляет собой процесс, с помощью которого ДНК существенно удваивается. Это важный процесс, происходящий внутри делящейся клетки.
В этой статье мы кратко рассмотрим структуру ДНК, точные этапы репликации ДНК (инициация, удлинение и завершение) и клинических последствий , которые могут произойти, когда что-то пойдет не так.
Структура ДНК
ДНК состоит из миллионов нуклеотидов. Это молекулы, состоящие из дезоксирибозного сахара с фосфатом и присоединенным к нему основанием (или азотистым основанием). Эти нуклеотиды прикреплены друг к другу цепями через фосфодиэфирные связи с образованием «сахарно-фосфатного остова». Образуется связь между третьим атомом углерода на сахаре дезоксирибозы одного нуклеотида (далее известном как 3 ’) и пятым атомом углерода другого сахара на следующем нуклеотиде (известном как 5’).
Примечание: 3 произносится как «три простых», а 5 »- как« пять простых ».
Две нити проходят в противоположных или антипараллельных направлениях друг к другу. Они прикреплены друг к другу по всей длине цепи через основания на каждом нуклеотиде. Есть 4 различных основания, связанных с ДНК; Цитозин, гуанин, аденин и тимин. В нормальных цепях ДНК цитозин связывается с гуанином, а аденин — с тимином. Две нити вместе образуют двойную спираль.
Рис. 1.0. Структура РНК и ДНК [/ caption]Этапы репликации ДНК
репликацию ДНК можно представить в виде трех стадий; Начало, удлинение, завершение
Инициирование
Синтез ДНКинициируется в определенных точках внутри цепи ДНК, известных как « origin », которые являются конкретными кодирующими областями. Эти источники являются мишенями для белков-инициаторов, которые продолжают привлекать больше белков, которые помогают процессу репликации, образуя комплекс репликации вокруг источника ДНК.Есть несколько сайтов происхождения, и когда начинается репликация ДНК, эти сайты обозначаются как r , вилки эпликации .
В репликационном комплексе находится фермент ДНК-геликаза , который раскручивает двойную спираль и обнажает каждую из двух цепей, так что их можно использовать в качестве матрицы для репликации. Он делает это путем гидролиза АТФ, используемого для образования связей между азотистыми основаниями, тем самым разрывая связь, удерживающую две цепи вместе.
ДНК-примаза — еще один фермент, который важен для репликации ДНК. Он синтезирует небольшой праймер РНК , который действует как «пусковой механизм» для ДНК-полимеразы . ДНК-полимераза — это фермент, который в конечном итоге отвечает за создание и расширение новых цепей ДНК.
Удлинение
После того, как ДНК-полимераза прикрепилась к исходным, разархивированным двум цепям ДНК (то есть цепям шаблона ), она может начать синтез новой ДНК в соответствии с шаблонами.Важно отметить, что ДНК-полимераза способна удлинить праймер только путем добавления свободных нуклеотидов к 3 ’концу .
Один из шаблонов читается в направлении от 3 ’к 5’, что означает, что новая нить будет сформирована в направлении от 5 ’к 3’ . Эта вновь сформированная нить называется ведущей нитью . Вдоль этой цепи ДНК-примазе необходимо синтезировать только праймер РНК один раз вначале, чтобы инициировать ДНК-полимеразу.Это связано с тем, что ДНК-полимераза способна удлинить новую цепь ДНК, считывая матрицу от 3 ‘до 5’, синтезируя в направлении от 5 ‘до 3’, как указано выше.
Однако другая цепочка-матрица (запаздывающая цепочка ) антипараллельна и, следовательно, считывается в направлении от 5 ’к 3’ . Непрерывный синтез ДНК, как в ведущей цепи , должен происходить в направлении от 3 ‘до 5’, что невозможно, поскольку мы не можем добавлять основания к 5 ‘концу. Вместо этого, когда спираль раскручивается, праймеры РНК добавляются к вновь экспонированным основаниям на запаздывающей цепи , и синтез ДНК происходит фрагментами, , но все еще в направлении от 5 ‘к 3’, как и раньше.Эти фрагменты известны как фрагменты казаки O .
Прекращение
Процесс расширения новых цепей ДНК продолжается до тех пор, пока либо не останется ни одной матрицы ДНК для репликации (т.е. в конце хромосомы), либо две репликационные вилки встретятся, а затем завершатся. Встреча двух вилок репликации не регулируется и происходит случайно по ходу хромосомы.
После завершения синтеза ДНК важно, чтобы вновь синтезированные цепи были связаны и стабилизировались.Что касается отстающей цепи, для этого необходимы два фермента; РНКаза H удаляет праймер РНК, который находится в начале каждого фрагмента Окадзаки, а ДНК-лигаза объединяет фрагменты вместе, чтобы создать одну полную цепь.
Рис. 2.0 — Схематическое изображение репликации ДНК [/ caption][старт-клиника]
Клиническая значимость — серповидноклеточная анемия
Серповидноклеточная анемия — это аутосомно-рецессивное заболевание, которое вызывается заменой одного основания , , при котором только одно основание заменяется другим.В некоторых случаях это может привести к «молчащей мутации», при которой не затрагивается весь ген, однако при таких заболеваниях, как серповидноклеточная анемия, это приводит к тому, что цепь кодирует другой белок.
В этом случае основание аденина заменяется на основание тимина в одном из генов, кодирующих гемоглобин; это приводит к замене глутаминовой кислоты на валин. Когда он транскрибируется в полипептидную цепь, свойства, которыми он обладает, радикально изменяются, поскольку глутаминовая кислота является гидрофильной, а валин гидрофобной.Эта гидрофобная область приводит к тому, что гемоглобин имеет ненормальную структуру, которая может вызывать закупорку капилляров, что приводит к ишемии и потенциально некрозу тканей и органов — это известно как вазоокклюзионный кризис .
Эти кризы обычно лечатся различными обезболивающими, включая опиоиды и НПВП, в зависимости от степени тяжести. Переливания красных кровяных телец могут потребоваться в экстренных случаях, например, если закупорка происходит в легких.
Рис. 3.0. Разница в структуре между нормальными эритроцитами и эритроцитами, пораженными серповидно-клеточной анемией. [/ caption][окончание клинической]
Самовоспроизводящиеся наноструктуры из ДНК
Реплицирующие сущности. Предоставлено: (c) Чонхун Ким и др. Nature Nanotechnology , DOI: 10.1038 / nnano.2015.87(Phys.org) — Возможно ли создание самовоспроизводящихся наноматериалов? Это могло быть, если бы мы позаимствовали строительные блоки природы.ДНК — это самовоспроизводящаяся молекула, в которой ее составные части, нуклеотиды, вступают в специфические химические взаимодействия, которые позволяют создавать самособирающиеся структуры. В биологических системах ДНК реплицируется с помощью белков. Однако Чон Хун Ким, Джунвье Ли, Сёго Хамада, Сатоши Мурата и Сунг Ха Парк из Университета Сунгюнкван и Университета Тохоку разработали управляемую самовоспроизводящуюся систему, не требующую белков. Их работа появилась в Nature Nanotechnology .
Чтобы понять, как работает этот самовоспроизводящийся процесс, важно знать его различные составляющие. Kim et al. разработали два Т-мотива ДНК, r 1 и r 2 , которые представляют собой двухцепочечную ДНК, состоящую из функциональных доменов, обозначенных альфа и бета, и «липких» концов в качестве точек соединения. Они также разработали мотив расширения. Двенадцать единиц мотива r 1 самостоятельно собираются в маленькое кольцо, R 1 , и двенадцать единиц r 2 плюс двенадцать дополнительных мотивов самостоятельно собираются в большее кольцо, R 2 .
Эти компоненты могут быть в двух разных состояниях: «удобренный» или «неоплодотворенный». Удобренные структуры содержат особенности, необходимые для воспроизведения. Оплодотворение происходит, когда одноцепочечный альфа- или бета-домен мотива r 1 или r 2 связывается с цепью, имеющей комплементарный альфа- или бета-домен. Это оставляет однониточный выступ или опору, идущую от кольца или от исходного рисунка. Зацепы указывают на то, что кольцо или мотив оплодотворены.
Эти опоры, отходящие от кольца ДНК, связываются с дополнительными цепями захватчиков. Когда это происходит, гибридная структура, состоящая из опоры для ног и вторгающейся нити, отрывается от исходного кольца, и в конечном итоге, когда эти части отламываются из-за миграции ветвей, они самостоятельно собираются в другое кольцо.
Этот процесс продолжается двумя разными путями репликации. Один путь растет экспоненциально. Другой путь растет согласно последовательности Фибоначчи.Конкретный выбранный путь зависит от того, какие вторгающиеся нити добавляются в систему.
Авторы подтвердили, что популяции колец ДНК росли благодаря этому процессу, опосредованному опорой, с помощью АСМ и исследований оптической плотности. Для исследований АСМ они взяли небольшой образец из каждой фазы и определили среднее количество колец, присутствующих в этой фазе. Данные по абсорбции были скорректированы для определения относительной концентрации колец в каждой фазе.
Они также подтвердили, что дочерние кольца были результатом отжига с одноцепочечными опорами исходного кольца, а не результатом самосборки остаточных мотивов ДНК в растворе с использованием гель-электрофореза и извлечения продуктов ДНК из каждой фазы.Отдельные фазы изучали с помощью АСМ, и вторгающиеся нити добавляли к раствору во время каждой из фаз, чтобы увидеть, образовались ли кольца.
Ким и др. продемонстрировали, что саморепликация в наномасштабе может происходить с использованием термодинамических свойств опосредованного зацепления смещения цепи и способности мотивов ДНК к самосборке. В этом исследовании синтетические Т-мотивы ДНК самоорганизуются в структуры, которые позволяют протекать последовательным реакциям. Это исследование демонстрирует возможность функционально программируемых самовоспроизводящихся наноструктур.
Общие принципы самосборки блоков ДНК
Дополнительная информация: «Саморепликация колец ДНК» Nature Nanotechnology , DOI: 10.1038 / nnano.2015.87
Аннотация
Биология предоставляет множество примеров самовоспроизводящихся машин, но искусственное создание таких сложных систем остается сложной задачей.В частности, хотя были разработаны простые искусственные самовоспроизводящиеся системы, включая деревянные блоки, магнитные системы, модульные роботы и синтетические молекулярные системы, такие кинематические самовоспроизводящиеся системы редки по сравнению с примерами теоретической клеточной саморепликации. Одна из основных причин этого — сложность, возникающая при попытке включить саморепликацию в физический носитель. В этом отношении ДНК является основным материалом-кандидатом для построения самовоспроизводящихся систем из-за ее способности к самосборке посредством молекулярного распознавания.Здесь мы показываем, что Т-мотивы ДНК, которые самособираются в кольцевые структуры, могут быть сконструированы для саморепликации посредством реакций смещения цепи, опосредованных пальцами ног. Собственная конструкция этих колец позволяет контролировать динамику популяции систем. Мы также анализируем схему репликации в универсальных рамках самовоспроизведения и выводим количественный показатель самовоспроизводимости колец.
© 2015 Phys.org
Ссылка : Самовоспроизводящиеся наноструктуры из ДНК (28 мая 2015 г.) получено 21 февраля 2021 г. с https: // физ.org / news / 2015-05-самовоспроизводящиеся-наноструктуры-dna.html
Этот документ защищен авторским правом. За исключением честных сделок с целью частного изучения или исследования, никакие часть может быть воспроизведена без письменного разрешения. Контент предоставляется только в информационных целях.
Репликативная геликаза в форме кольца окружает двухцепочечную ДНК во время раскрутки | Исследование нуклеиновых кислот
Абстрактные
Кольцеобразные репликативные геликазы являются гексамерными и играют ключевую роль в репликации клеточной ДНК.Несмотря на их важность, наше понимание механизма раскручивания репликативных геликаз далеко от совершенства. Вирус папилломы крупного рогатого скота E1 — одна из самых известных модельных систем для репликативных геликаз. E1 — это многофункциональный инициатор, который распознает и расплавляет вирусное происхождение и раскручивает ДНК. Здесь мы изучаем механизм раскрутки E1 на уровне одной молекулы с помощью магнитного пинцета. Результат показывает, что E1 как отдельный гексамер представляет собой плохо процессивную геликазу с низкой скоростью раскручивания.Натяжение цепей ДНК препятствует раскручиванию, указывая на то, что геликаза сильно взаимодействует с обеими цепями ДНК в месте соединения. Исследуя взаимодействие при высокой силе (26–30 пН), мы обнаружили, что E1 окружает дцДНК. Сравнивая с конструкцией E1 без домена связывания ДНК, мы предлагаем два возможных режима окружения E1 во время активного раскручивания.
ВВЕДЕНИЕ
Репликация ДНК — наиболее важный биологический процесс для размножения жизни.Репликативные геликазы играют ключевую роль в раскручивании ДНК, чтобы инициировать репликацию и возглавлять ряд белков, необходимых для последующего синтеза ДНК. Из-за важности репликативных геликаз их структура и механизм раскручивания стали предметом интенсивных исследований. Вирус папилломы крупного рогатого скота E1 — одна из самых известных модельных систем для изучения репликативных геликаз в эукариотических клетках с большим Т-антигеном SV40 (LTag) (1,2). E1 — это многофункциональный инициаторный белок, который необходим для репликации вирусных эписом (3).Во время репликации E1 распознает и плавит вирусный источник ( ori ) и раскручивает ДНК в направлении 3′-5 ‘(2–4). В отличие от прокариотических репликативных геликаз, E1 и LTag имеют тот же домен AAA + аденозинтрифосфатазы (АТФаза), что и репликативные геликазы архей и эукариот (1). E1 — гомогексамер, состоящий исключительно из идентичных мономеров, тогда как репликативные геликазы эукариот представляют собой комплексы, состоящие из различных типов субъединиц (5). Хотя E2, хорошо известный партнер взаимодействия E1, необходим для набора E1 в ori в vivo , он является незаменимым в в анализах раскручивания vitro (6,7).Более простая структура этого раскручивающегося комплекса делает E1 полезным в качестве модельной системы для репликативных геликаз эукариот.
Домен E1 делится на N-концевую регуляторную область, ДНК-связывающий домен (DBD) и геликазный домен (HD) (1,2). N-концевой регуляторный домен обеспечивает регуляцию в vivo . Он содержит несколько мотивов последовательности, необходимых для нуклео-цитоплазматического перемещения E1. DBD распознает последовательность ori и связывается с ней.HD дополнительно делится на домен олигомеризации (OD) и домен АТФазы, содержащий гибкий С-концевой хвост. HD индуцирует сборку в олигомерную форму и раскручивает ДНК путем гидролиза АТФ. E1 собирается либо в двойной гексамер (DH) в присутствии последовательностей или и АТФ, либо в одиночный гексамер (SH) в присутствии оцДНК (8-10). И DH, и SH обладают активностью разматывания. В отличие от DH, SH не обладал специфичностью последовательности. Эффективность разматывания дцДНК без ori , как правило, очень низка, поскольку не может образоваться активная DH (11).Чтобы раскрутить дцДНК без или , требуется 3′-хвостовой оцДНК, на который может загружаться SH (9,10). Активное раскручивание и транслокация происходят только в сочетании с гидролизом АТФ (12).
Все репликативные геликазы для репликации клеточной ДНК принимают форму гексамерного кольца (5,13–14). Хотя составляющие структурные мотивы различаются в разных гексамерных геликазах, гомология в трехмерной (3D) структурной архитектуре предполагает сходный процесс раскручивания (1,15-16). Грубо говоря, процесс раскрутки инициируется сборкой гексамерной геликазы на ori .Пройдя конформационный переход в активную форму, он начинает раскручивать ДНК. Биохимические и структурные исследования гексамерных геликаз предоставили обширную информацию о подробном механизме; однако некоторые ключевые аспекты все еще остаются неуловимыми. Большинство гексамерных геликаз архей и эукариот собираются как DHs в точке ori (5,11). E1 и LTag могут быть собраны как SH или DH в зависимости от ДНК-субстрата (5,11). Хотя DH, по-видимому, является основной формой при сборке, неясно, сохраняется ли DH или разделяется на SHs во время фазы активного раскручивания в vivo .Фактически, формы DH и SH сосуществовали в ЭМ-структуре разматывающе-активного комплекса LTag (17). Однако эксперимент Yardimci и др. с растяжением потока. ясно продемонстрировали, что гексамеры LTag разделяются во время репликации ДНК, визуализируя разделение репликационных вилок (18). Доказательства относительно E1 все еще неубедительны, потому что экспериментальные методы, которые могут контролировать олигомерные состояния в реальном времени, еще не применялись, и, в отличие от LTag, только форма DH наблюдалась в аналогичных исследованиях EM (19).Независимо от того, разделяются ли гексамерные единицы, существует единый консенсус для механизмов разматывания в единой гексамерной единице. Считается, что большинство гексамерных геликаз раскручивают ДНК посредством модели стерического исключения (SE) (5,15,20). В модели SE геликаза охватывает одну цепь и отделяет ее от другой цепи SE. Важный прорыв в понимании механизма раскручивания E1 был обеспечен рентгеновской структурой E1, сокристаллизованной с оцДНК, описанной в 2006 г. (21).Структура показала, что оцДНК действительно окружена геликазой и проходит через центральный канал, что сильно поддерживает модель SE. Результаты анализа одномолекулярного FRET также позволяют предположить, что E1 следует модели SE (22). Модель SE рассматривает цепь, по которой геликаза не перемещается [нетранслирующая цепь (NTL)], как пассивный компонент, возникающий в результате раскручивания. Однако возможность того, что нить NTL взаимодействует с геликазами и, таким образом, влияет на раскручивание, была увеличена за последнее десятилетие (23-26).Типичным примером является Saccharolobus solfataricus MCM (SsoMCM), структура которого выявила, что нить NTL была намотана вокруг внешней стороны геликазы (27). Этот режим разматывания получил название модели стерического исключения и упаковки (SEW) (20,28). С другой стороны, известно, что центральные каналы некоторых гексамерных геликаз слишком широки для размещения только оцДНК, что означает, что дцДНК может проходить через центральный канал (29,30). Недавно группа О’Доннелл предложила модифицированную модель SE (MSE) после того, как обнаружила, что некоторая дцДНК в репликационной вилке проникает в центральный канал эукариотического комплекса CMG (20,31).Это открытие указывает на то, что цепь NTL не только взаимодействует с геликазой, но также окружена геликазой вместе с перемещающейся цепью (TL). Эта модель отличается от модели SE тем, что дуплекс разделяется внутри геликазы. Кристаллическая структура E1 с оцДНК (21) была получена с использованием конструкции E1 без N-концевой половины, тогда как аналогичная структура, окружающая дцДНК, была обнаружена при использовании полноразмерного E1 с субстратами репликационной вилки (32). Кроме того, нить NTL, которая вошла в геликазу, оказалась вытесненной из бокового канала на стенке геликазы.Фактически, гексамерные геликазы, как известно, обладают положительно заряженными боковыми каналами, которые могут использоваться в качестве выхода экструзии для оцДНК (16,29,32–34). Langston et al. относится к режиму разматывания, в котором оцДНК экструдируется из бокового канала, как к модели экструзии бокового канала (SCE) (35). Обе модели MSE и SCE полагают, что дцДНК окружает, однако нить NTL в модели MSE вытесняется из центрального канала, а не из бокового канала. Хотя возможность механизма раскручивания, отличного от модели SE, признается, она не была экспериментально подтверждена для E1, особенно во время активного раскручивания ДНК.
Здесь мы исследуем механизм раскручивания E1 на уровне одной молекулы с помощью магнитного пинцета. Результаты, полученные с субстратами, имитирующими репликационную вилку, показывают, что SH-форма E1 имеет слабую раскручивающую активность. Сила отрицательно влияет на активность разматывания, указывая на то, что существует взаимодействие между геликазой и цепями ДНК. При исследовании взаимодействия мы обнаружили, что дцДНК окружена E1. Сравнивая результаты E1HD без DBD, мы предлагаем две возможные структуры E1, которые различаются в основном своим окружающим поведением.
МАТЕРИАЛЫ И МЕТОДЫ
Очистка от вируса папилломы E1
E1 (DBD + HD, 128–605) и E1HD (только HD, 308–605) из бычьего папилломавируса типа 1 (рис. 1A) были экспрессированы через Escherichia coli штамм BL21 (DE3) в виде N-концевых слияний GST и очищают аффинной хроматографией на глутатион-агарозе. Часть GST расщепляли тромбином и очищали ионообменной хроматографией. Подробная очистка описана (10,36).
Рисунок 1.
Характерная динамика раскрутки E1. ( A ) Использовали два вида конструкций E1. Один — E1 (остаток 128–605), который состоит из DBD и HD. Другой — E1HD (остатки 308–605), который содержит только HD. ( B ) Схема теста магнитного пинцета для контроля разматывания E1. В то время как HP притягивается парой магнитов с силой ниже F расстегивает , мономеры E1 и ATP-Mg 2+ смываются в камеру.E1 собирается в активную гексамерную форму, а затем начинает раскручивать HP. Результирующее увеличение удлинения (Δz) отслеживается и преобразуется в количество размотанных пар оснований. ( C ) Типичная кривая разматывания E1 на HP50bp при 5 пН. E1 может полностью раскрутить HP50bp. После достижения максимума HP постепенно перематывался из-за перемещения E1 за вершину шпильки. ( D ) E1 продемонстрировал разнообразную динамику от монотонной одиночной размотки (две верхние панели) до повторяющейся размотки (две нижние панели).Между двумя разматывающими движениями более преобладает повторяющееся движение, составляющее 65%. При повторяющемся движении E1 двигалась назад через резкое проскальзывание или постепенную перемотку назад. ( E ) Чтобы оценить процессивность раскрутки E1, мы провели эксперимент с более длинным HP (HP1kbp). Число пар оснований, развернутых E1 в течение 500 с, широко распределялось от нескольких десятков (две верхние панели) до более чем 100 п.о. (нижняя панель) при 5 пН. Однако максимальное количество развернутых пар оснований, как правило, было ниже 180 п.н.
Рисунок 1.
Характерная динамика раскрутки E1. ( A ) Использовали два вида конструкций E1. Один — E1 (остаток 128–605), который состоит из DBD и HD. Другой — E1HD (остатки 308–605), который содержит только HD. ( B ) Схема теста магнитного пинцета для контроля разматывания E1. В то время как HP притягивается парой магнитов с силой ниже F расстегивает , мономеры E1 и ATP-Mg 2+ смываются в камеру.E1 собирается в активную гексамерную форму, а затем начинает раскручивать HP. Результирующее увеличение удлинения (Δz) отслеживается и преобразуется в количество размотанных пар оснований. ( C ) Типичная кривая разматывания E1 на HP50bp при 5 пН. E1 может полностью раскрутить HP50bp. После достижения максимума HP постепенно перематывался из-за перемещения E1 за вершину шпильки. ( D ) E1 продемонстрировал разнообразную динамику от монотонной одиночной размотки (две верхние панели) до повторяющейся размотки (две нижние панели).Между двумя разматывающими движениями более преобладает повторяющееся движение, составляющее 65%. При повторяющемся движении E1 двигалась назад через резкое проскальзывание или постепенную перемотку назад. ( E ) Чтобы оценить процессивность раскрутки E1, мы провели эксперимент с более длинным HP (HP1kbp). Число пар оснований, развернутых E1 в течение 500 с, широко распределялось от нескольких десятков (две верхние панели) до более чем 100 п.о. (нижняя панель) при 5 пН. Однако максимальное количество развернутых пар оснований, как правило, было ниже 180 п.н.
Приготовление конструкций шпильки ДНК
ДНК-шпилька-конструкция (HP) была приготовлена, как описано ранее (37). В экспериментах использовали два HP со стеблями 50 и 1038 п.н. (HP50bp и HP1kbp соответственно). Каждый HP состоит из ручки дигоксигенина, вставки и стержня шпильки (дополнительный рисунок S1). Ручка дигоксигенина предназначена для иммобилизации на поверхности стекла, покрытой антидигоксигенином. Вставка между рукояткой дигоксигенина и шпилькой сердечника предотвращает слишком близкое приближение магнитных шариков к поверхности стекла.Центральная шпилька содержит одноцепочечный участок (dT 30 ) для загрузки геликазы и дуплексный стержень, который геликаза раскручивает. На 5′-конце стержневой шпильки три молекулы биотина помечены для связывания с покрытыми стрептавидином магнитными шариками, а на другом конце имеется выступ из 10 оснований для лигирования со вставкой. Ручка дигоксигенина и вставка были приготовлены путем расщепления фрагментов полимеразной цепной реакции (ПЦР) эндонуклеазами рестрикции. В случае ручки дигоксигенина ПЦР проводилась в присутствии дигоксигенина-dUTP (Roche Diagnostics).Шпилька ядра HP50bp была сделана отжигом двух олигонуклеотидов (Integrated DNA Technologies, Coralville, IA, USA). Содержание GC в стержне HP50bp составляет 32%. Центральную шпильку HP1kbp получали лигированием предварительно отожженного олигонуклеотида, фрагмента ПЦР размером ~ 1 т.п.н. и самоотжигаемой петли. Содержание GC в стержне HP1kbp составляет 47%. Вся конструкция была собрана путем лигирования трех частей с соответствующим дополнительным выступом и очищена. Все последовательности олигонуклеотидов приведены в дополнительной таблице S1.
Магнитный пинцет для анализа
Проточные кюветы были собраны с двумя покровными стеклами и листом парафильма и герметизированы путем плавления парафильма. Верхнее стекло имело два отверстия для замены буфера, а нижнее стекло функционализировали нитроцеллюлозой (0,1% мас. / Об. В амилацетате). Гранулы полистирола диаметром 3,2 мкм (Invitrogen), которые служили эталоном для коррекции дрейфа, вводили в проточную ячейку и иммобилизовали на поверхности путем нагревания при 100 ° C в течение 1 мин.Затем проточные кюветы инкубировали со 100 мкг / мл анти-дигоксигенина (Roche Diagnostics) при 4 ° C в течение ночи. Для уменьшения неспецифического связывания смесь дополнительно инкубировали с 10 мг / мл бычьего сывороточного альбумина (BSA) (New England Biolabs) в течение не менее 1 часа при комнатной температуре. Проточные кюветы промывали 1 × фосфатно-солевым буфером после каждой стадии инкубации. Покрытые стрептавидином магнитные шарики диаметром 2,8 мкм (M280, Invitrogen) смешивали с HP и инкубировали в течение 10 мин. Несвязавшаяся ДНК была удалена путем выпадения.Комплекс бусина-шпилька ДНК был введен в проточную ячейку. После инкубации в течение 5 минут несвязавшиеся шарики тщательно промывали связывающим буфером E1 (буфер E1BS; 50 мМ HEPES (pH 7,6), 100 мМ NaCl, 5 мМ MgCl 2 , 1 мМ DTT, 0,005% Triton X-100. , 0,1 мг / мл БСА).
Анализ магнитного пинцета проводился на самодельной установке, которая была сконструирована так же, как описанная в предыдущей статье (38). Пара магнитов, изготовленных из NdFeB (W-05-N50-G, Supermagnete), была установлена на линейном моторизованном столике (M-126, Physik Instrumente) и размещена над проточной ячейкой.Сила контролировалась изменением расстояния от проточной ячейки. Для получения дифракционной картины шарики освещали видимым светодиодным источником света (CR5111A-WY, Roithner Lasertechnik). Свет, собранный через масляно-иммерсионный объектив (Olympus, N.A. 1.25), отображался с помощью камеры CCD (EoSens CL MC1362, Mikrotron) при 120 Гц. Позиции x и y шарика были получены из центра дифракционного кольца. Положение Z определяли путем сопоставления измеренной дифракционной картины с серией эталонных картин, созданных при смене линзы объектива.Положение линзы объектива контролировалось пьезоэлектрическим столиком (P-721, Physik Instrumente). Дрейф был скорректирован с использованием положения эталонных шариков. Сила калибровалась как функция расстояния между магнитами и проточной ячейкой для молекул дцДНК размером 11 т.п.н.
Эксперименты по размотке Helicase
Перед экспериментами по раскручиванию все молекулы шпильки ДНК были протестированы, чтобы определить, произошел ли резкий переход при расстегивании молнии около силы расстегивания, F unzip (дополнительный рисунок S2).Были измерены только молекулы, демонстрирующие правильное поведение при расстегивании (дополнительный рисунок S3). Раскручивание было инициировано промывкой буфера E1BS, содержащего геликазу E1 (140 нМ) и АТФ (2 мМ), в молекулы HP (рис. 1B). Иногда мы вводили E1 без АТФ, чтобы исследовать только процесс сборки. Концентрация E1 поддерживалась как можно более низкой, чтобы избежать множественного связывания белков и неспецифической ассоциации с ДНК. Растяжение бусинки ДНК регистрировали при силе 5–10 пН. Увеличение удлинения при раскручивании было преобразовано в количество размотанных пар оснований с использованием эластичного свойства оцДНК, рассчитанного с помощью расширяемой модели FJC (см. Дополнительный рисунок S4).Для расчета скорости раскрутки (UW) необработанные данные были отфильтрованы через скользящее среднее с 120 точками окна. Для расчета были выбраны только периоды, в которых раскрутка была непрерывной. Мы определили процессивность по максимальному количеству пар оснований, которое E1 раскручивает за ~ 500 с. В эксперименте по измерению активности транслокации вводили E1 или E1HD, в то время как HP был механически расстегнут. После снижения усилия были записаны следы. Когда геликаза собирается в оцДНК мимо петли HP, HP постепенно восстанавливается после транслокации геликазы.Мы периодически разархивировали HP, чтобы сравнить скорость постепенного повторного архивирования и транслокации (TR). Скорость TR рассчитывалась по наклону вниз. Чтобы исследовать взаимодействие между нитями ДНК и E1, мы многократно увеличивали силу до очень высокого значения (26 или 30 пН), записывая следы раскрутки при 10 пН. Время действия большой силы составляло ~ 10 с.
РЕЗУЛЬТАТЫ
Неоднородная динамика разматывания геликазы Е1
Чтобы инициировать раскручивание, E1 вводили в молекулы HP с АТФ с силой ниже F расстегиваем (рис. 1B).Если присутствует ori , E1 сначала связывается с ori как двойной тример (DT). После локального плавления или DT преобразуется в активный DH и начинает раскручивание (39). Чтобы избежать вмешательства со стороны плавления ori и сфокусировать активность SH, мы использовали HP, не содержащий BPV ori . Без ori , E1 будет предпочтительно собираться вокруг одноцепочечной области HP в форму SH. Известно, что E1 SH занимает около 30 оснований (12).Следовательно, каждый HP мог разместить только один гексамер. Когда E1 начал раскручивать HP, расширение постепенно увеличивалось, как показано на рисунке 1C. Без АТФ такого увеличения не наблюдалось. Сначала мы провели эксперименты с E1 и HP50bp, чтобы определить общие характеристики динамики разматывания. Репрезентативные следы, полученные при 5 пН, показаны на рисунке 1D. Динамика разматывания была разнообразной, от одиночной монотонной размотки (две верхние панели на рисунке 1D) до повторяющейся размотки (две нижние панели на рисунке 1D).Поскольку повторное раскручивание продолжалось после удаления остаточной геликазы, это происходило не из-за диссоциации и реассоциации E1. На второй панели рисунка 1D дуплекс был полностью размотан на первой фазе разматывания. E1 непрерывно перемещается к 5′-концу, и дуплекс постепенно повторно застегивается позади E1. В этом случае раскрутка не возобновлялась и завершалась одним монотонным событием. Иногда E1 возвращался в исходное положение до полного разматывания и возобновлял размотку.Затем возникла повторяющаяся размотка, как показано на двух нижних панелях фиг.1D. Интересно, что повторяющееся раскручивание прекращается, когда E1 полностью разматывает дуплекс и достигает положения на 5′-стороне посредством непрерывной транслокации. Это может быть связано с тем, что E1 на стороне 5 ‘не мог сразу вернуться в исходное положение. Эта разнообразная и гетерогенная динамика согласуется с предыдущим результатом анализа FRET одной молекулы (22). Для количественного анализа динамики рассчитывались скорости ВВ на склонах, во время которых разматывание было непрерывным.Скорость UW при 5 пН составила 0,90 ± 0,57 пар оснований / с. Хотя скорость UW была заметно ниже, чем у прокариотических гексамерных геликаз, таких как T7 gp4, T4 gp41 и DnaB (30-50 п.н. / с) (40-42), она была сопоставима с таковой LTag (1-2 п.н. / с). ) (43,44). При повторном раскручивании E1 двигался назад либо постепенно, либо внезапно. Такое обратное движение часто наблюдается, когда геликазы полностью раскручивают шпильки ДНК, потому что соединения дц / оцДНК, к которым стабильно прикреплены геликазы, исчезают (45). Однако движение E1 назад произошло независимо от достижения полного раскручивания (см. Первую и третью панели на рисунке 1D).Это поведение похоже на поведение геликаз RecQ, которые продолжают раскручиваться, регрессировать назад и возобновлять (37,45). Процессивность измеряли с более длинным HP (HP1kbp). Хотя он был несколько широко распределен, как показано на графиках на Рисунке 1E, процессивность E1 не превышала 180 п.н., что было намного ниже, чем у LTag с аналогичной скоростью UW. При аналогичной экспериментальной конфигурации LTag может раскручивать несколько пар оснований без диссоциации (43,44). Известно также, что MCM архей способны раскручиваться по крайней мере на 500 п.н. (46,47).Этот результат показывает, что E1, как SH, является плохой геликазой.
Сила препятствует разматыванию E1
Чтобы понять механизм разматывания, мы провели эксперименты с различными силами. Типичные следы, полученные с HP50bp, представлены на рисунке 2A. Скорости UW и процессивность, полученные при различных силах, суммированы на фиг. 2B и C. Геликазы подразделяются на активные или пассивные геликазы в зависимости от механизма, с помощью которого они разделяют дуплекс.Пассивные геликазы раскручивают ДНК, просто захватывая временно расплавленные соединения посредством тепловых колебаний, тогда как активные геликазы раскручивают ДНК, понижая энергетический барьер. Большинство гексамерных геликаз являются пассивными или частично пассивными геликазами (48,49). В пассивных геликазах скорость UW значительно увеличивается по мере увеличения силы, поскольку сила способствует плавлению соединения. Чтобы определить, какой тип E1, мы исследовали раскручивание под действием различных сил. Неожиданно мы обнаружили противоположную тенденцию: скорость UW уменьшалась с увеличением силы (рис. 2B).Скорость UW при 10 пН составляла 0,23 ± 0,16 пар оснований / с, что составляет четверть от таковой при 5 пН (0,90 ± 0,57 пар оснований / с). Большие погрешности в данных были связаны с неоднородностью динамики. Учитывая зависимость скорости ВВ от силы, E1 нельзя было отнести к пассивной геликазе. Затем мы проверили, является ли E1 активной геликазой. Согласно критерию Manosas et al. (48), геликазы, скорость UW которых превышает четверть скорости TR, считаются активными геликазами. Скорость TR, определенная по нисходящему наклону на рис. 3A и B, составила 4.19 ± 1,88 п.н. / с при 10 пН. Поскольку скорость UW была ниже четверти скорости TR при всех силах, E1 также не подходил для активной модели. В общем, скорость UW увеличивается с увеличением силы, хотя чувствительность различается в зависимости от того, являются ли они активными геликазами или пассивными геликазами. Например, режим I E1HD, который будет обсуждаться позже, показывает поведение активных геликаз. Скорость UW и процессивность E1HD Mode I умеренно увеличивались по мере увеличения силы, как показано на Рисунках 2B и C.Хотя данные только по E1 казались несколько разбросанными из-за большой ошибки, отрицательный эффект силы был очевиден при сравнении данных с данными режима E1HD. Аналогичная тенденция наблюдалась также в скорости RW (дополнительный рисунок S5A) и в целом. время для полной размотки (дополнительный рисунок S5B). Тот факт, что сила, приложенная к двум цепям ДНК в противоположном направлении, препятствовала раскручиванию, указывает на то, что геликаза взаимодействует с цепью NTL, которая не окружена геликазой.Попытка классифицировать E1 как активную или пассивную геликазу не удалась, вероятно, потому, что модель не учитывает такое взаимодействие. Об отрицательном эффекте силы также сообщалось для гексамерных геликаз T4 gp41 и DnaB, которые пришли к такому же выводу (26,42).
Рисунок 2.
Влияние силы на разматывающую активность E1. ( A ) Типичные следы раскручивания E1, полученные с HP50bp при различных усилиях. ( B ) Скорость разматывания (UW) E1 для HP50bp (сплошные серые квадраты) и HP1kbp (сплошные синие треугольники) измеряли как функцию силы.Для сравнения были нанесены на график скорости UW E1HD для HP1kbp (сплошные оранжевые кружки для режима I и сплошные розовые кружки для режима II). Для расчета скорости UW было проанализировано в общей сложности 205 наклонов от 96 молекул HP, и для каждой точки данных использовалось более восьми событий. Степень UW E1 уменьшалась по мере увеличения силы независимо от длины HP, что указывает на отрицательный эффект силы. В частности, скорость UW для HP50bp составляла 0,90 ± 0,57 bp / s (стандартное отклонение для N = 20 событий) при 5 пН и 0.23 ± 0,16 п.н. / с (стандартное отклонение для N = 11 событий) при 10 пН. Скорости UW E1HD Mode I (сплошные оранжевые кружки) были больше, чем у E1 во всех диапазонах сил. В отличие от E1, скорость UW режима E1HD Mode I увеличивалась с увеличением силы, хотя чувствительность была не очень высокой из-за активного характера геликазы. Скорость UW режима E1HD Mode II (сплошной розовый кружок), измеренная при 10 пН, была аналогична таковой для E1. ( C ) Максимальные развернутые пары оснований в течение заданного времени (процессивности) E1 и E1HD на HP1kbp были собраны при различных силах для 29 и 14 молекул HP соответственно.Процессивность E1 была плохой при всех силах, тогда как E1HD была очень высокой и дополнительно усиливалась увеличением силы, достигая 1 т.п.н. при 10 пН.
Рис. 2.
Влияние силы на разматывающую активность E1. ( A ) Типичные следы раскручивания E1, полученные с HP50bp при различных усилиях. ( B ) Скорость разматывания (UW) E1 для HP50bp (сплошные серые квадраты) и HP1kbp (сплошные синие треугольники) измеряли как функцию силы. Для сравнения были нанесены на график скорости UW E1HD для HP1kbp (сплошные оранжевые кружки для режима I и сплошные розовые кружки для режима II).Для расчета скорости UW было проанализировано в общей сложности 205 наклонов от 96 молекул HP, и для каждой точки данных использовалось более восьми событий. Степень UW E1 уменьшалась по мере увеличения силы независимо от длины HP, что указывает на отрицательный эффект силы. В частности, скорость UW для HP50bp составляла 0,90 ± 0,57 пар оснований / с (стандартное отклонение для N = 20 событий) при 5 пН и 0,23 ± 0,16 пар оснований / с (стандартное отклонение для N = 11 событий) при 10 пН. Скорости UW E1HD Mode I (сплошные оранжевые кружки) были больше, чем у E1 во всех диапазонах сил.В отличие от E1, скорость UW режима E1HD Mode I увеличивалась с увеличением силы, хотя чувствительность была не очень высокой из-за активного характера геликазы. Скорость UW режима E1HD Mode II (сплошной розовый кружок), измеренная при 10 пН, была аналогична таковой для E1. ( C ) Максимальные развернутые пары оснований в течение заданного времени (процессивности) E1 и E1HD на HP1kbp были собраны при различных силах для 29 и 14 молекул HP соответственно. Процессивность E1 была плохой при всех силах, тогда как E1HD была очень высокой и дополнительно усиливалась увеличением силы, достигая 1 т.п.н. при 10 пН.
Рисунок 3.
Транслокация E1 и E1HD на одноцепочечной ДНК. ( A ) Для отслеживания активности транслокации вводили E1, а HP1kbp механически распаковывали. Дождавшись сборки E1 на одноцепочечной ДНК, мы снизили усилие ниже F распаковать и записали следы. Когда E1 был связан с ДНК за петлей шпильки, наблюдалось постепенное уменьшение удлинения из-за повторного сжатия после TL-геликазы, как в фазе (I).Чтобы сравнить постепенное повторное сжатие (I) с транслокацией по оцДНК (II), мы периодически распаковывали HP в течение ~ 10 с. Смещение по z (Δz) в течение периода распаковки является показателем транслокации E1 вдоль оцДНК. Скорости TR рассчитывались либо из наклонов вниз в фазе (I), либо из | Δz | / Δt в фазе (II). Скорости TR, рассчитанные по фазам (I) и (II), были практически идентичны. Следы транслокации ( B ) E1 и ( C ) E1HD. Скорости TR E1 и E1HD, рассчитанные по трассам, составили 4.19 ± 1,88 и 4,47 ± 1,29 п.н. / с соответственно, что согласуется с ошибкой.
Рисунок 3.
Транслокация E1 и E1HD на одноцепочечной ДНК. ( A ) Для отслеживания активности транслокации вводили E1, а HP1kbp механически распаковывали. Дождавшись сборки E1 на одноцепочечной ДНК, мы снизили усилие ниже F распаковать и записали следы. Когда E1 был связан с ДНК за петлей шпильки, наблюдалось постепенное уменьшение удлинения из-за повторного сжатия после TL-геликазы, как в фазе (I).Чтобы сравнить постепенное повторное сжатие (I) с транслокацией по оцДНК (II), мы периодически распаковывали HP в течение ~ 10 с. Смещение по z (Δz) в течение периода распаковки является показателем транслокации E1 вдоль оцДНК. Скорости TR рассчитывались либо из наклонов вниз в фазе (I), либо из | Δz | / Δt в фазе (II). Скорости TR, рассчитанные по фазам (I) и (II), были практически идентичны. Следы транслокации ( B ) E1 и ( C ) E1HD. Скорости TR E1 и E1HD, рассчитанные по трассам, составили 4.19 ± 1,88 и 4,47 ± 1,29 п.н. / с соответственно, что согласуется с ошибкой.
Двухцепочечная ДНК окружена E1
По отрицательному влиянию силы на раскручивание мы обнаружили, что существует взаимодействие между нитью NTL и геликазой. Чтобы выяснить природу взаимодействия, мы приложили большое усилие к HP при записи следов при 10 пН. В этом эксперименте вводили только E1, а позже добавляли АТФ для отдельного мониторинга процессов сборки и разматывания.Без E1 голый HP (HP1kbp) был быстро распакован с более высоким усилием, чем F распаковал и повторно застегнут с меньшим усилием (дополнительный рисунок S3). Однако после того, как был введен E1, HP никогда не разархивировался, даже при 26 пН, что намного выше, чем у F unzip . HP оставался закрытым, в то время как активное разматывание происходило путем добавления АТФ (см. Рисунок 4A). Тот факт, что E1 полностью блокировал разделение соединения, указывает на то, что E1 прочно связывается с нитью NTL в дополнение к окруженной нити TL.Незначительное увеличение растяжения при высокой силе было связано с эластичным удлинением части дцДНК, вставленной между стержневой шпилькой и дигоксигениновой ручкой (см. Дополнительный рисунок S1). Как упоминалось ранее, модель SEW, основанная на результатах SsoMCM, предполагает, что размотанная нить NTL оборачивается вокруг геликазы. В SsoMCM было обнаружено, что упаковка удерживается за счет электростатического взаимодействия, которое, как предполагалось, было слабым, чтобы позволить размотанной нити скользить по внешней стороне геликазы (27). Если нить NTL связана путем обертывания, как в модели SEW, она не выдержит такой большой силы.Другое основание для этого рассуждения состоит в том, что сильное захватное поведение E1 наблюдалось без АТФ и, следовательно, без существенного раскручивания. Тогда единственной доступной оцДНК было 5 оснований на 5′-конце ядра HP, которое было слишком коротким, чтобы обернуться вокруг геликазы. Следовательно, маловероятно, что нить NTL была захвачена геликазой через внешнюю обертку. В модели MSE, упомянутой в разделе «Введение», одна цепь введенной дцДНК (цепь NTL) повторно экструдируется от входа (31).Возможно взаимодействие между нитью NTL и внутренней поверхностью геликазы. Подобно модели SEW, взаимодействие не должно быть сильным для нити NTL, чтобы постоянно ускользать от геликазы. В крио-ЭМ геликазы CMG, которая следует модели MSE, экструдированная нить не может быть визуализирована из-за высокой подвижности (31). Высокомобильная нить NTL несовместима с сильным взаимодействием, наблюдаемым в этой работе. Следовательно, остающаяся возможность состоит в том, что нить NTL окружена E1 и экструдируется через боковой канал на стенке геликазы, как было предложено Chaban et al. (32). Как только нить NTL окружена таким образом, механическая сила не может легко вытащить ее из геликазы без разрыва кольца геликазы.
Рисунок 4.
Окружение дцДНК E1 наблюдается во время периодического приложения большой силы (> F unzip ). ( A ) Большое усилие (26 пН) часто прикладывалось в течение 10 секунд при записи следов разматывания при 10 пН для HP1kbp. Перед инъекцией E1 HP был полностью расстегнут с большим усилием обратимым способом.Однако после инъекции E1 HP не мог быть распакован с большим усилием без АТФ и оставался закрытым после добавления АТФ. Этот сильный захват обеих цепей HP указывает на окружение дцДНК E1. ( B ) Все возможные пути экструзии нити NTL из E1 во время разматывания. Путь (i) предшествует проникновению в геликазу, тогда как пути (ii) и (iii) происходят из побочного канала между уровнями DBD и OD и перед уровнем АТФазы, соответственно. Путь (iv) показывает, что нить NTL проходит через центральный канал вместе с нитью TL.Путь (i) был исключен на основании наблюдения за поведением сильного захвата, а путь (iv) был исключен из-за отсутствия активности E1 по транслокации дцДНК. Следовательно, наиболее вероятный путь будет либо (ii), либо (iii), которые отмечены розовым цветом. ( C ) Репрезентативный след первой моды (Режим I) между двумя различными режимами разматывания, наблюдаемыми в экспериментах с E1HD. В отличие от E1, E1HD не помешал полностью распаковать HP при большом усилии. Показатель UW составил 2,72 ± 0.91 б.п. / с, что выше, чем у E1. E1HD в Mode Я смог процессивно раскрутить HP1kbp без пауз. ( D ) След E1HD во втором режиме (Mode II) наблюдался сначала и преобразовался в Mode I позже. Режим II E1HD показал очень похожие характеристики с E1, такие как низкая скорость UW и сильное сцепление с большим усилием. Иногда во время приложения большой силы наблюдались внезапные скачки в растяжении, как показано в середине кривой. Мы подозреваем, что ДНК была вытащена из геликазы из-за большой силы.Интересно, что E1HD в состоянии Mode II изменился на состояние Mode I, показывая быстрое разматывание и полное разархивирование при многократном приложении чуть более высокой силы 30 пН.
Рисунок 4.
Окружение дцДНК E1, наблюдаемое во время периодического приложения большой силы (> F распаковать ). ( A ) Большое усилие (26 пН) часто прикладывалось в течение 10 секунд при записи следов разматывания при 10 пН для HP1kbp. Перед инъекцией E1 HP был полностью расстегнут с большим усилием обратимым способом.Однако после инъекции E1 HP не мог быть распакован с большим усилием без АТФ и оставался закрытым после добавления АТФ. Этот сильный захват обеих цепей HP указывает на окружение дцДНК E1. ( B ) Все возможные пути экструзии нити NTL из E1 во время разматывания. Путь (i) предшествует проникновению в геликазу, тогда как пути (ii) и (iii) происходят из побочного канала между уровнями DBD и OD и перед уровнем АТФазы, соответственно. Путь (iv) показывает, что нить NTL проходит через центральный канал вместе с нитью TL.Путь (i) был исключен на основании наблюдения за поведением сильного захвата, а путь (iv) был исключен из-за отсутствия активности E1 по транслокации дцДНК. Следовательно, наиболее вероятный путь будет либо (ii), либо (iii), которые отмечены розовым цветом. ( C ) Репрезентативный след первой моды (Режим I) между двумя различными режимами разматывания, наблюдаемыми в экспериментах с E1HD. В отличие от E1, E1HD не помешал полностью распаковать HP при большом усилии. Показатель UW составил 2,72 ± 0.91 б.п. / с, что выше, чем у E1. E1HD в Mode Я смог процессивно раскрутить HP1kbp без пауз. ( D ) След E1HD во втором режиме (Mode II) наблюдался сначала и преобразовался в Mode I позже. Режим II E1HD показал очень похожие характеристики с E1, такие как низкая скорость UW и сильное сцепление с большим усилием. Иногда во время приложения большой силы наблюдались внезапные скачки в растяжении, как показано в середине кривой. Мы подозреваем, что ДНК была вытащена из геликазы из-за большой силы.Интересно, что E1HD в состоянии Mode II изменился на состояние Mode I, показывая быстрое разматывание и полное разархивирование при многократном приложении чуть более высокой силы 30 пН.
Два режима разматывания E1HD раскрывают механизм разматывания E1
Поведение сильного захвата указывает на то, что дцДНК была окружена E1 и что нить NTL была выдавлена через боковой канал. Согласно исследованию FRET одной молекулы, соединение дц / оцДНК находится на N-концевой стороне геликазы (22).Поскольку наша конструкция E1 не содержала N-концевого регуляторного домена, дцДНК должна входить в уровень DBD. Хотя позиция входа казалась ясной, положение, из которого выдавливается нить NTL, все еще оставалось неуловимым. Все возможные пути экструзии показаны на рисунке 4B. Повторное выдавливание на входе, как в модели MSE, можно исключить, как указано выше. Сильный захват можно наблюдать, когда нить NTL проходит через центральный канал и выходит на стороне АТФазы (рис. 4B, путь (iv)).Хотя некоторые гексамерные геликазы обладают активностью транслокации дцДНК (50,51), для E1 об этом не сообщалось. Кроме того, если обе цепи выходят со стороны АТФазы, раскручивание не может наблюдаться в нашем эксперименте, потому что ДНК немедленно регибридизируется вслед за геликазой. Следовательно, маловероятно, что нить NTL выходит со стороны АТФазы. Учитывая общую трехмерную структуру, позиция, из которой нить NTL может быть выдавлена, является побочным каналом между доменами DBD и ATPase (рис. 4B).
Для дальнейшего изучения окружения дцДНК мы провели измерения для E1HD, лишенного DBD. Когда только E1HD вводили в HP1kbp без АТФ, сильное сцепление наблюдали при высокой концентрации E1HD 1,3 мкМ (дополнительный рисунок S6). К сожалению, активного раскручивания после добавления АТФ мы не наблюдали. Поэтому не было уверенности в том, что E1HD, сильно захвативший HP, был компетентной формой раскрутки. Однако, когда E1HD вводили одновременно с АТФ, происходило активное раскручивание, как показано на рисунке 4C.Динамика разматывания и сцепление E1HD были более сложными, чем у E1. После тщательного анализа мы обнаружили, что два разных режима раскрутки сосуществуют в E1HD с одинаковой вероятностью. Мы разделили их на «режим I» и «режим II» в зависимости от характеристик захвата. В режиме I HP подверглась обратимому распаковке при 26 пН (рис. 4C). Такое поведение указывает на отсутствие сильного взаимодействия между нитью NTL и E1HD. Чтобы дополнительно охарактеризовать режим I, мы проанализировали скорости UW и TR, а также процессивность.Скорость UW режима I составляла 2,72 ± 0,91 пар оснований / с при 10 пН, что в 10 раз выше, чем у E1 (рисунок 2B). Кроме того, режим I был гораздо более процессорным, позволяя полностью раскрутить HP1kbp (рисунок 2C). Скорости TR, рассчитанные из наклонов, показанных на рисунке 3C, составили 4,47 ± 1,29 п.п. / с при 10 пН. Сравнивая скорости UW и TR, режим I можно классифицировать как активную геликазу. Исчезновение сильного захвата при удалении DBD означает, что нить NTL была окружена DBD в E1. Как ни странно, E1HD часто демонстрировал другую динамику, Mode II.В режиме II HP не распаковывался при 26 пН (рис. 4D), что указывает на окружение дцДНК. Хотя скорость UW (0,69 ± 0,41 пар оснований / с) была немного выше (рис. 2B), общие характеристики режима II были очень похожи на характеристики E1. Сравнивая результаты E1HD Mode II и E1, мы пришли к совершенно разному выводу, что влияние DBD на раскручивание было минимальным, и нить NTL все еще была окружена E1HD. Наблюдение за тем, что удаление DBD привело как к режиму I, так и к режиму II, подразумевает наличие двух различных конформаций в E1, которые различаются, главным образом, кольцевой структурой.Интересно, что мы обнаружили, что HP, демонстрирующая динамику режима II, может преобразоваться в режим I, теряя свое сильное сцепление с нитью NTL, когда 30 пН применялись повторно (рис. 4D). Этот результат демонстрирует, что две моды произошли от видов с одинаковым химическим составом. Основываясь на наших результатах, казалось, что высокая сила высвобождает нить NTL из геликазы за счет частичного разрыва кольца геликазы.
ОБСУЖДЕНИЕ
Используя метод магнитного пинцета, мы исследовали динамику раскручивания E1 для субстратов ДНК репликационной вилки.Было обнаружено, что E1, как SH, является плохой геликазой с низкой скоростью раскрутки и низкой процессивностью. В частности, процессивность E1 составляла самое большее ~ 180 п.н., что резко контрастирует с высоко процессивным LTag, который может непрерывно раскручивать ~ 2 п.н. (43,44). Для эффективной репликации вирусного эписома размером 6-7 т.п.н., вероятно, необходимы другие факторы для усиления активности раскручивания. E1 собран с вирусным ori в форме DH. Димеризация в DH может быть таким стимулирующим фактором. Это согласуется с недавними наблюдениями о том, что изменение олигомеризации является важным регуляторным механизмом для модуляции активности и функциональности геликаз (49).Поскольку SH не обладает специфичностью последовательности, он может случайным образом раскручивать ДНК везде, где есть разветвленное соединение. Слабая раскручивающая активность E1 SH, по-видимому, подавляет раскручивание, происходящее в сайтах, отличных от или , без надлежащей координации двух гексамеров E1 и родственных белков. Это было бы особенно важно, поскольку на латентной стадии количество вирусных эписом составляет не более 100 копий (52), что чрезвычайно мало по сравнению с клеточной ДНК. Димеризация LTag, как известно, важна для репликации ДНК (53,54).Однако сильная процессивность LTag как формы SH подразумевает, что димеризация не является предпосылкой для устойчивой репликации ДНК после того, как ori плавится и устанавливаются две вилки (18). Помимо димеризации, взаимодействие с другими белками может быть стимулирующим фактором, как показано в геликазе DEAD-бокса elF4A и WRN (37,55). E2 — типичный белок, который взаимодействует с E1. Хотя E2 необходим для рекрутирования E1 в ori в vivo , он сам по себе не увеличивает раскручивающую активность (7,56).Белок репликации А (RPA), эукариотический связывающий белок оцДНК, взаимодействует со многими геликазами, включая E1 (57). Ярким примером является геликаза WRN, процессивность которой резко увеличивается с помощью RPA (37). Однако известно, что E1 не стимулируется RPA (58). Вместо этого E1 и RPA конкурируют за связывание оцДНК. Для эффективной репликации ДНК активность репликативной геликазы, как правило, связана с активностью ДНК-полимеразы (59). Ассоциация с полимеразой может увеличивать процессивность репликативной геликазы.Фактически, процессивность полимеразы ϵ, как известно, стимулируется E1 (60). Однако об обратном, а именно о стимуляции E1 полимеразами, пока не сообщалось.
Исходя из эффекта отрицательной силы, мы предположили, что существует взаимодействие между нитью NTL и геликазой. Изучая взаимодействие, мы обнаружили, что обе нити ДНК были окружены геликазой. Хотя известно, что E1 DH окружает dsDNA во время плавления ori (11,16), неясно, остается ли dsDNA окруженной после того, как инициируется активное раскручивание.ЭМ-структура полноразмерного E1 по Chaban et al. (32) показали, что короткий участок дцДНК в репликационной вилке окружен геликазой. Авторы также предположили, что окруженная нить NTL экструдируется через боковой канал между уровнями DBD и OD. На основе структуры они предсказали, что E1 будет следовать модели SE, если N-концевая половина будет удалена (рис. 5A, слева). Наш эксперимент с использованием E1HD дал смешанные результаты, половина из которых совпадала с предсказанием, а другая половина — нет.В E1HD было два режима разматывания: режим I демонстрировал обратимое разархивирование HP и быстрое разматывание, а режим II показал характеристики, аналогичные E1. Отсутствие сильного сцепления в Режиме I, похоже, согласуется с предсказанием Чабана и др. Следовательно, разумно предположить, что E1 адаптирует структуру, предложенную авторами (Рисунок 5B, слева). Напротив, Mode II, который напоминает E1, указывает, что DBD лишь незначительно влияет на динамику раскрутки и что dsDNA окружена другими уровнями доменов в E1HD.Первый N-уровень, который дцДНК должна входить в E1HD, — это уровень OD (22). Однако, согласно кристаллической структуре апо-E1, диаметр канала на уровне OD составляет ∼13 Å (61), что мало для размещения дцДНК ∼20 Å. Недавние структуры LTag и MCM с высоким разрешением с дцДНК выявили несколько интересных особенностей (62,63). В частности, следует отметить, что LTag был способен окружать дцДНК по всему центральному каналу, несмотря на небольшой диаметр канала (14-19 Å) на уровне АТФазы. Неожиданно оказалось, что узкий канал сжимает и деформирует dsDNA, нарушая спаривание оснований Watson-Crick.Мы предполагаем, что OD-уровень E1HD может окружать дцДНК посредством аналогичного механизма; Домены OD, собранные вокруг дцДНК, частично плавят дцДНК, образуя жесткий воротник. Поскольку нить NTL не будет полностью проходить через центральный канал, как объяснено в разделе «Результаты», разумно предположить, что расплавленная нить NTL экструдируется через боковой канал перед уровнями АТФазы (рис. 5A, справа). Таким образом, вторая возможная структура E1 на основе E1HD Mode II будет следующей.Сначала дцДНК окружена слоями DBD и OD, частично плавится на уровне OD, а затем одна из расплавленных цепей экструдируется через боковой канал перед уровнем АТФазы (рис. 5B, справа). Сосуществование режима I и режима II предполагает, что E1 может адаптировать любую из двух структур, описанных выше. Однако мы думаем, что второй вариант (рис. 5B, справа) более вероятен, учитывая близкое сходство в динамике раскрутки между E1 и Mode II E1HD. Другое основание для нашего предположения состоит в том, что если дцДНК была окружена только DBD, она не смогла бы противостоять таким высоким силам, потому что межсубъединичное взаимодействие между шестью DBD, как известно, довольно слабое (64).Однако способность DBD связывать ДНК может помочь относительно небольшим доменам OD окружать дцДНК, сближая две нити ДНК. Следовательно, удаление DBD увеличило бы популяцию в режиме I.
Рисунок 5.
Ожидаемые модели размотки E1 и E1HD. ( A ) Две модели размотки E1HD, основанные на характеристиках двух различных режимов разматывания, наблюдаемых в этом исследовании. В структуре, основанной на режиме I (слева), нить NTL исключена перед геликазой.Поскольку воротник OD является жестким и имеет небольшой диаметр полости, разматывание, вероятно, будет следовать модели SE, в которой уровень OD исключает нить NTL. Напротив, в структуре, основанной на режиме II (справа), дцДНК окружена уровнем OD, а нить NTL экструдируется из бокового канала перед уровнем АТФазы. При сборке гексамера дцДНК, по-видимому, частично расплавляется за счет сильной силы олигомеризации между OD. Эта модель отличается от модели SE тем, что происходит разделение дуплексов внутри геликазы.( B ) Предлагаемые размоточные модели E1. Поведение сильного захвата, наблюдаемое в E1, указывает на то, что по крайней мере один уровень домена должен окружать дцДНК. Рассматривая окружение дцДНК и структуры E1HD, мы предлагаем две различные раскручивающиеся структуры E1. Основное различие между структурами заключается в том, где выдавливается нить NTL. В левой структуре нить NTL экструдируется перед входом в уровень OD. В правой структуре нить NTL выдавливается после уровня OD. Мы думаем, что правильная модель более вероятна, учитывая схожесть динамики E1 и E1HD Mode II.
Рисунок 5.
Ожидаемые модели размотки E1 и E1HD. ( A ) Две модели размотки E1HD, основанные на характеристиках двух различных режимов разматывания, наблюдаемых в этом исследовании. В структуре, основанной на режиме I (слева), нить NTL исключена перед геликазой. Поскольку воротник OD является жестким и имеет небольшой диаметр полости, разматывание, вероятно, будет следовать модели SE, в которой уровень OD исключает нить NTL. Напротив, в структуре, основанной на режиме II (справа), дцДНК окружена уровнем OD, а нить NTL экструдируется из бокового канала перед уровнем АТФазы.При сборке гексамера дцДНК, по-видимому, частично расплавляется за счет сильной силы олигомеризации между OD. Эта модель отличается от модели SE тем, что происходит разделение дуплексов внутри геликазы. ( B ) Предлагаемые размоточные модели E1. Поведение сильного захвата, наблюдаемое в E1, указывает на то, что по крайней мере один уровень домена должен окружать дцДНК. Рассматривая окружение дцДНК и структуры E1HD, мы предлагаем две различные раскручивающиеся структуры E1. Основное различие между структурами заключается в том, где выдавливается нить NTL.В левой структуре нить NTL экструдируется перед входом в уровень OD. В правой структуре нить NTL выдавливается после уровня OD. Мы думаем, что правильная модель более вероятна, учитывая схожесть динамики E1 и E1HD Mode II.
Следует отметить, что раскрутка была быстрой и процессивной, когда оцДНК была окружена (E1HD Mode I), тогда как она была медленной и плохо процессорной, когда дцДНК была окружена (E1 и E1HD Mode II). Однако различия в скорости и процессивности UW могут быть не такими большими в в vivo , где сила отсутствует или низкая, учитывая тенденцию, показанную на рисунках 2B и C.Биохимические анализы показали, что процессивность E1 и E1HD значительно различалась; E1HD развернутый длиной более 1 кб, тогда как E1 развернут не более 0,2 кб (12,22). Режим I E1HD, вероятно, ответственен за сильную процессивность, наблюдаемую с E1HD. Как показали биохимические анализы, проведенные без силы, E1 был хуже по активности, чем E1HD Mode I, даже при нулевой силе. Слабая раскручивающая активность E1, по-видимому, связана с трудностью, с которой дцДНК проходит через узкий центральный канал, особенно на уровне OD.Окружающая дцДНК может действовать как регуляторный механизм для замедления активности раскручивания. Мы предполагаем, что частичное открытие N-яруса будет переключателем для перехода из режима плохой размотки в режим надежной размотки, как это происходит при силе 30 пН, разрывая кольцо геликазы и вытягивая из него нить. Сообщалось, что Mcm10 в значительной степени стимулирует раскручивающую активность комплекса CMG, и в то же время он позволяет комплексу CMG, который остановился в блоке на нити NTL, обходить блок (65).Основываясь на наших результатах, мы предполагаем, что Mcm10 способствовал раскручивающей активности комплекса CMG, переводя его из режима медленного окружения дцДНК в режим быстрого SE. Как и геликазы RecQ, E1 часто многократно раскручивался, хотя степень повторения была намного ниже, чем у RecQ. RecQ катализирует различные вторичные структуры ДНК в мультимерной форме. Повторяющееся раскручивание геликаз RecQ было приписано синхронизации с другими субъединицами в ожидании связывания других субъединиц с ДНК (37).Аналогично, действие E1 может быть действием по гармонизации с другим гексамером или комплексом репликации. Независимо от того, какой E1 принимает из двух предложенных нами структур, дцДНК разделяется внутри геликазы, что не согласуется с общепринятой моделью SE. Мы полагаем, что окружение дцДНК не ограничивается вирусными геликазами, потому что структурные данные с высоким разрешением и уточненные биохимические анализы эукариотических гексамерных геликаз также побудили к модификации существующих моделей SE.
ДОПОЛНИТЕЛЬНЫЕ ДАННЫЕ
Дополнительные данные доступны в NAR Online.
БЛАГОДАРНОСТИ
Проф. Бон Хён Чон посвятил всю свою жизнь тому, чтобы быть страстным ведущим исследователем. Авторы хотели бы посвятить эту статью профессору Бон Хён Чонгу, который скончался 24 февраля 2016 года в разгар этого проекта. Кроме того, мы благодарим профессора Арне Стенлунда (Лаборатория Колд-Спринг-Харбор) за предоставленные белки E1 и E1HD и доктора Сунг-Дже Ли за ценные советы по предварительным экспериментам.
ФИНАНСИРОВАНИЕ
Разработка платформенных технологий для программы инновационных медицинских измерений [KRISS-2019-GP2019-0013]; Программа исследовательских инициатив [KRIBB]; Программа конвергенции НИОКР [CAP-14-3-KRISS, NST]. Финансирование платы за открытый доступ: Программа конвергенции НИОКР [CAP-14-3-KRISS, NST].
Заявление о конфликте интересов . Ничего не объявлено.
ССЫЛКИ
1.Hickman
A.B.
,Dyda
F.
Связывание и раскручивание: вирусные геликазы SF3
.Curr. Мнение. Struct. Биол.
2005
;15
:77
—85
.2.Bergvall
M.
,Melendy
T.
,Archambault
J.
Белки E1
.Вирусология
.2013
;445
:35
—56
.3.Seo
Ю.С.
,Мюллер
Ф.
,Lusky
M.
,Hurwitz
J.
Белок E1, кодируемый вирусом папилломы крупного рогатого скота (BPV), содержит несколько активностей, необходимых для репликации ДНК BPV
.Proc. Natl. Акад. Sci. США
1993
;90
:702
—706
.4.Ян
L.
,Mohr
I.
,Fouts
E.
,Lim
D.A.
,Нохайле
М.
,Botchan
M.
Белок E1 вируса папилломы крупного рогатого скота 1 является АТФ-зависимой ДНК-геликазой
.Proc. Natl. Акад. Sci. США
1993
;90
:5086
—5090
. 5.O’Donnell
M.E.
,Li
H.
Кольцевые гексамерные геликазы, которые функционируют в ответвлениях репликации ДНК
.Nat. Struct. Мол. Биол.
2018
;25
:122
—130
.6.Ян
L.
,Li
R.
,Mohr
I.J.
,Clark
R.
,Botchan
M.R.
Активация репликации BPV-1 in vitro фактором транскрипции E2
.Природа
.1991
;353
:628
—632
.7.Gopalakrishnan
V.
,Khan
S.A.
Белка E1 вируса папилломы человека типа 1a достаточно для инициации репликации вирусной ДНК
.Proc. Natl. Акад. Sci. США
1994
;91
:9597
—9601
.8.Fouts
E.T.
,Yu
X.
,Egelman
E.H.
,Botchan
M.R.
Биохимический и электронный микроскопический анализ изображений гексамерной геликазы E1
.J. Biol. Chem.
1999
;274
:4447
—4458
.9.Titolo
S.
,Pelletier
A.
,Pulichino
A.-M.
,Brault
K.
,Wardrop
E.
,Белый
P.W.
,Cordingley
M.G.
,Archambault
J.
Идентификация доменов геликазы E1 вируса папилломы человека типа 11, участвующих в олигомеризации и связывании с источником вируса
.J. Virol.
2000
;74
:7349
—7361
.10.Sedman
J.
,Stenlund
A.
Белок папилломавируса E1 образует ДНК-зависимый гексамерный комплекс с АТФазной и ДНК-геликазной активностями
.J. Virol.
1998
;72
:6893
—6897
.11.Schuck
S.
,Stenlund
A.
Сборка двойной гексамерной геликазы
.Мол. Ячейка
.2005
;20
:377
—389
.12.Castella
S.
,Burgin
D.
,Sanders
C.M.
Роль гидролиза АТФ в ДНК-транслоказной активности геликазы Е1 вируса папилломы крупного рогатого скота (BPV-1)
.Nucleic Acids Res.
2006
;34
:3731
—3741
.13.Patel
S.S.
,Picha
K.M.
Строение и функции гексамерных геликаз
.Annu. Rev. Biochem.
2000
;69
:651
—697
.14.Тракселис
МА
Структурные механизмы нагружения, сборки и разматывания гексамерной геликазы [вариант 1; экспертная оценка: 3 одобрены]
.F1000Res.
2016
;5
:111
.15.Brewster
A.S.
,Чен
X.S.
Изучение функционального механизма MCM: уроки, извлеченные из архейного комплекса MCM
.Крит. Rev. Biochem. Мол. Биол.
2010
;45
:243
—256
. 16.Stenlund
A.
Инициирование репликации ДНК: уроки вирусных белков-инициаторов
.Nat. Преподобный Мол. Cell Biol.
2003
;4
:777
—785
.17.Wessel
R.
,Schweizer
J.
,Stahl
H.
Геликаза ДНК Т-антигена обезьяньего вируса 40 представляет собой гексамер, который образует бинарный комплекс во время двунаправленного раскручивания вируса. ориджин репликации ДНК
.J. Virol.
1992
;66
:804
—815
. 18.Yardimci
H.
,Wang
X.
,Loveland
A.B.
,Tappin
I.
,Rudner
D.Z.
,Hurwitz
J.
,Oijen
A.M.V.
,Walter
J.C.
Обход белкового барьера репликативной ДНК-геликазой
.Природа
.2012
;492
:205
—209
.19.Лин
B.Y.
,Махов
А.М.
,Гриффит
Дж.D.
,Брокер
T.R.
,Чау
L.T.
Белки-шапероны отменяют ингибирование репликативной геликазы Е1 вируса папилломы человека (ВПЧ) белком
ВПЧ E2.Мол. Cell Biol.
2002
;22
:6592
—6604
.20.Trakselis
M.A.
,Seidman
M.M.
,Брош
Р.М.
Механистическое понимание того, как геликаза CMG способствует репликации, преодолевая препятствия на пути ДНК
.Ремонт ДНК
.2017
;55
:76
—82
. 21.Enemark
E.J.
,Джошуа-Тор
L.
Механизм транслокации ДНК в репликативной гексамерной геликазе
.Природа
.2006
;442
:270
—275
. 22.Ли
S.-J.
,Syed
S.
,Enemark
E.J.
,Schuck
S.
,Stenlund
A.
,Ha
T.
,Joshua-Tor
L.
Динамический взгляд на раскручивание ДНК репликативной геликазой
.Proc. Natl Acad. Sci. США
2014
;111
:E827
—E835
. 23.Фу
Ю.В.
,Yardimci
H.
,Long
D.T.
,Ho
T.V.
,Guainazzi
A.
,Bermudez
V.P.
,Hurwitz
J.
,Oijen
A.v.
,Schärer
O.D.
,Уолтер
J.C.
Избирательный обход блокиратора отстающей цепи репликативной ДНК-геликазой эукариот
.Ячейка
.2011
;146
:931
—941
. 24.Ротенберг
E.
,Trakselis
M.A.
,Bell
S.D.
,Ha
T.
Специфичность раздвоенного субстрата MCM включает динамическое взаимодействие с 5′-хвостом
.J. Biol. Chem.
2007
;282
:34229
—34234
. 25.Rabhi
M.
,Tuma
R.
,Boudvillain
M.
Ремоделирование РНК с помощью гексамерных РНК-геликаз
.RNA Biol.
2010
;7
:655
—666
. 26.Рибек
Н.
,Салех
О.А.
Раскручиванию ДНК кольцевой геликазой Т4 gp41 препятствует натяжение окклюзированной цепи
.PLoS One
.2013
;8
:e79237
.27.Graham
B.W.
,Schauer
G.D.
,Leuba
S.H.
,Trakselis
M.A.
Стерическое исключение и оборачивание исключенной цепи ДНК происходит вдоль дискретных внешних путей связывания во время раскручивания геликазы MCM
.Nucleic Acids Res.
2011
;39
:6585
—6595
. 28.Карни
S.M.
,Trakselis
M.A.
Исключенная цепь ДНК важна для раскручивания гексамерной геликазы
.Методы
.2016
;108
:79
—91
,29.Li
D.
,Zhao
R.
,Lilyestrom
W.
,Gai
D.
,Zhang
R.
,DeCaprio J. 9000
,Fanning
E.
,Jochimiak
A.
,Szakonyi
G.
,Chen
X.S.
Структура репликативной геликазы онкопротеина SV40 антигена большой опухоли
.Природа
.2003
;423
:512
—518
. 30.Cuesta
I.
,Núñez-Ramírez
R.
,Scheres
S.H.W.
,Гаи
Д.
,Чен
X.S.
,Fanning
E.
,Carazo
J.M.
Конформационные перестройки большого Т-антигена SV40 во время событий ранней репликации
.J. Mol. Биол.
2010
;397
:1276
—1286
. 31.Георгеску
R.
,юаней
Z.
,Bai
L.
,Santos
R.d.L.A.
,Вс
Дж.
,Чжан
Д.
,Юрьева
О.
,Li
H.
,O’Donnell
M.E.
Структура эукариотической геликазы CMG на вилке репликации и последствия для архитектуры реплисомы и инициации ориджина
.Proc. Natl. Акад. Sci. США
2017
;114
:E697
—E706
.32.Чабан
Y.
,Stead
J.A.
,Рыженкова
К.
,Велан
Ф.
,Ламбер
E.P.
,Antson
A.
,Sanders
C.M.
,Орлова
Е.В.
Структурная основа разделения цепей ДНК гексамерной репликативной геликазой
.Nucleic Acids Res.
2015
;43
:8551
—8563
. 33.Брюстер
A.S.
,Ван
г.
,Ю.
X.
,Гринлиф
W.B.
,Carazo
J.M.
,Tjajadi
M.
,Klein
M.G.
,Чен
X.S.
Кристаллическая структура почти полноразмерной архейной MCM: функциональные идеи для гексамерной геликазы AAA +
.Proc. Natl. Акад. Sci. США
2008
;105
:20191
—20196
. 34.Li
N.
,Zhai
Y.
,Zhang
Y.
,Li
W.
,Yang
M.
,Lei
J.
,B. Tye 9 -000 K3
.
,Gao
N.
Структура эукариотического комплекса MCM при 3,8 Å
.Природа
.2015
;524
:186
—191
.35.Лэнгстон
Л.
,О’Доннелл
М.
Действие CMG со специфичными для цепи блоками ДНК поддерживает режим внутреннего раскручивания для эукариотической репликативной геликазы
.Элиф
.2017
;6
:e23449
,36.Sedman
T.
,Sedman
J.
,Stenlund
A.
Связывание белков E1 и E2 с ориджином репликации вируса папилломы крупного рогатого скота
.J. Virol.
1997
;71
:2887
—2896
.37.Lee
M.
,Shin
S.
,Uhm
H.
,Hong
H.
,Kirk
J.
,Hyun
K. ,
Kulikowicz
T.
,Kim
J.
,Ahn
B.
,Bohr
VA
et al. .Множественные RPA превращают белок синдрома WRN в супергеликазу
.Nucleic Acids Res.
2018
;46
:4689
—4698
.38.Brutzer
H.
,Luzzietti
N.
,Klaue
D.
,Seidel
R.
Энергетика при переходе сверхспирали ДНК
.Biophys. J.
2010
;98
:1267
—1276
.39.Schuck
S.
,Stenlund
A.
Механистический анализ локального плавления ori и сборки геликазы с помощью белка
вируса папилломы E1.Мол. Ячейка
.2011
;43
:776
—787
.40.Lionnet
T.
,Spiering
M.M.
,Бенкович
S.J.
,Bensimon
D.
,Croquette
V.
Наблюдение в реальном времени за геликазой бактериофага T4 gp41 обнаруживает механизм разматывания
.Proc. Natl. Акад. Sci. США
2007
;104
:19790
—19795
.41.Johnson
D.S.
,Bai
L.
,Smith
B.Y.
,Patel
S.S.
,Wang
M.D.
Исследования одиночных молекул показывают динамику раскручивания ДНК кольцевой геликазой Т7
.Ячейка
.2007
;129
:1299
–1309
.42.Ribeck
N.
,Kaplan
D.L.
,Bruck
I.
,Saleh
O.A.
Активность геликазы DnaB модулируется геометрией ДНК и силой
.Biophys. J.
2010
;99
:2170
—2179
. 43.Клауэ
D.
Раскручивание ДНК геликазами, исследованное на уровне одиночной молекулы
.2012
; Кандидатская диссертацияТУ Дрезден
Физический факультет
.44.Burnham
D.R.
,Косе
H.B.
,Hoyle
R.B.
,Yardimci
H.
Механизм раскручивания ДНК эукариотической репликативной геликазой
.Nat. Commun.
2019
;10
:2159
.45.Klaue
D.
,Kobbe
D.
,Kemmerich
F.
,Kozikowska
A.
,Puchta
H.
,Seidel
R.
Антагонистическая активность RecQ-геликаз
в отношении чувствительности к вилкам и контроля переключения цепей.Nat. Commun.
2013
;4
:2024
.46.Чонг
J.P.J.
,Хаяси
М.К.
,Саймон
М.Н.
,Сюй
Р.-М.
,Stillman
B.
Поддерживающий белок минихромосомы архей с двойным гексамером представляет собой АТФ-зависимую ДНК-геликазу
.Proc. Natl. Акад. Sci. США
2000
;97
:1530
—1535
. 47.Шехтер
D.F.
,Инь
C.Y.
,Gautier
J.
Внутренняя активность ДНК-хеликазы methanobacterium thermoautotrophicum ΔH минихромосомный поддерживающий белок
.J. Biol. Chem.
2000
;275
:15049
—15059
. 48.Маносас
М.
,Xi
X.G.
,Bensimon
D.
,Croquette
V.
Активный и пассивный механизмы геликасов
.Nucleic Acids Res.
2010
;38
:5518
—5526
. 49.Patel
S.S.
,Donmez
I.
Механизмы геликасов
.J. Biol. Chem.
2006
;281
:18265
—18268
.50.Каплан
D.L.
,O’Donnell
M.
DnaB вызывает миграцию ветвей ДНК и вытесняет белки, окружая две нити ДНК
.Мол. Ячейка
.2002
;10
:647
—657
. 51.Шин
J.-H.
,Jiang
Y.
,Grabowski
B.
,Hurwitz
J.
,Kelman
Z.
Требования к субстрату для транслокации дуплексной ДНК с помощью поддерживающих хеликаз минихромосом эукариальных и архей
.J. Biol. Chem.
2003
;278
:49053
—49062
,52.Mecsas
J.
,Sugden
B.
Репликация плазмид, полученных из вируса папилломы крупного рогатого скота типа 1 и вируса Эпштейна-Барра, в клетках в культуре
.Annu. Rev. Cell Biol.
1987
;3
:87
—108
. 53.Weisshart
K.
,Taneja
P.
,Jenne
A.
,Herbig
U.
,Simmons
DT
000 Fanning
,000 Fanning области антигена 40 Т обезьяньего вируса определяют кооперативность сборки двойного гексамера на вирусном ориджине репликации ДНК и способствуют гексамерным взаимодействиям во время двунаправленного ориджина раскручивания ДНК
.J. Virol.
1999
;73
:2201
—2211
. 54.Sreekumar
K.R.
,Prack
A.E.
,Winters
D.R.
,Барбаро
Б.А.
,Bullock
P.A.
Источник ядра обезьяньего вируса 40 содержит два отдельных модуля последовательности, которые поддерживают сборку двойного гексамера Т-антигена
.J. Virol.
2000
;74
:8589
—8600
.55.Garcia-Garcia
C.
,Frieda
K.L.
,Феоктистова
K.
,Fraser
C.S.
,Block
S.M.
Фактор-зависимая процессивность геликазы eIF4A DEAD-box человека
.Наука
.2015
;348
:1486
—1488
. 56.Bonne-Andrea
C.
,Santucci
S.
,Clertant
P.
Белок E1 вируса папилломы крупного рогатого скота сам по себе может эффективно управлять множеством циклов синтеза ДНК in vitro
.J. Virol.
1995
;69
:3201
—3205
. 57.Фаннинг
Э.
,Климович
В.
,Нагер
А.Р.
Динамическая модель функции репликационного протеина А (RPA) в путях обработки ДНК
.Nucleic Acids Res.
2006
;34
:4126
—4137
. 58.Лоо
Ю.М.
,Melendy
T.
Рекрутирование белка репликации A белком E1 вируса папилломы и модуляция одноцепочечной ДНК
.J. Virol.
2004
;78
:1605
—1615
. 59.Patel
S.S.
,Pandey
M.
,Nandakumar
D.
Динамическое соединение между двигателями репликации ДНК: гексамерная геликаза, ДНК-полимераза и примаза
.Curr. Мнение. Chem. Биол.
2011
;15
:595
—605
.60.Chojnacki
M.
,Melendy
T.
Геликаза E1 ДНК вируса папилломы человека связывает, стимулирует и придает процессивность клеточной ДНК-полимеразе epsilon
.Nucleic Acids Res.
2018
;46
:229
—241
.61.Шлифовальные машины
C.M.
,Ковалевский
О.В.
,Сизов
Д.
,Лебедев
А.А.
,Исупов
М.Н.
,Антсон
А.А.
Сборка геликазы вируса папилломы E1 поддерживает асимметричное состояние в отсутствие ДНК и кофакторов нуклеотидов
.Nucleic Acids Res.
2007
;35
:6451
—6457
.62.Gai
D.
,Wang
D.
,Li
S.-X.
,Чен
X.S.
Структура большой гексамерной геликазы Т Т SV40 в комплексе с ДНК богатого АТ происхождения
.Элиф
.2016
;5
:e18129
.63.Али
F.A.
,Дуглас
М.E.
,Locke
J.
,Pye
V.E.
,Nans
A.
,Diffley
J.F.X.
,Коста
A.
Крио-ЭМ структура лицензированного ориджина репликации ДНК
.Nat. Commun.
2017
;8
:2241
.64.Enemark
E.
Кристаллическая структура ДНК-связывающего домена белка инициации репликации E1 из вируса папилломы
.Мол. Ячейка
.2000
;6
:149
—158
0,65.Лэнгстон
L.D.
,Mayle
R.
,Schauer
G.D.
,Yurieva
O.
,Zhang
D.
,Yao
N.Y.
, 9000,O’Donnell
M.

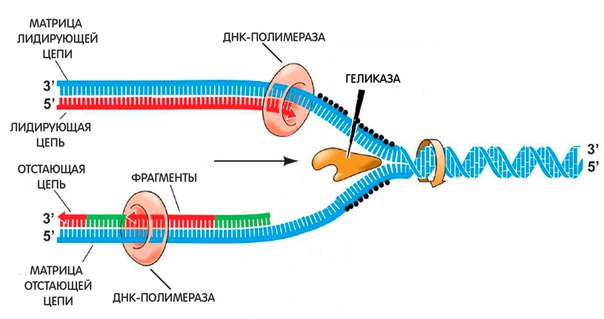

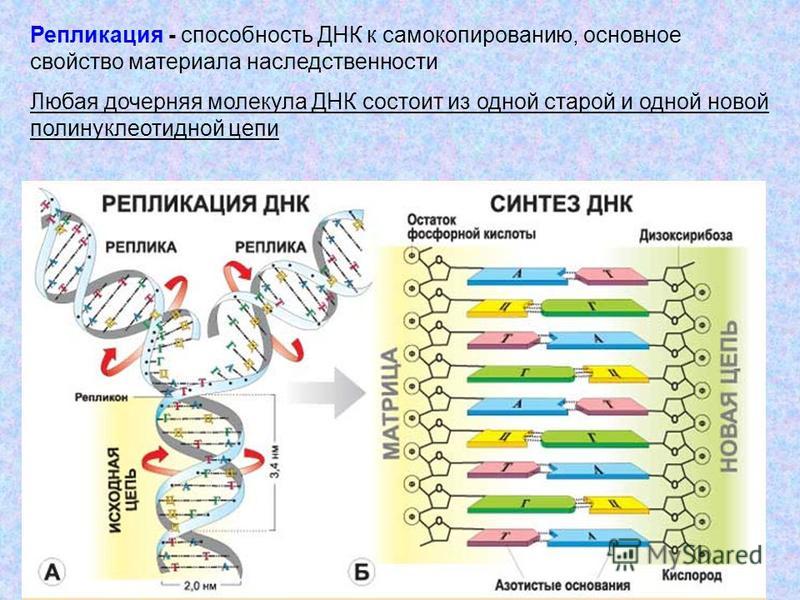
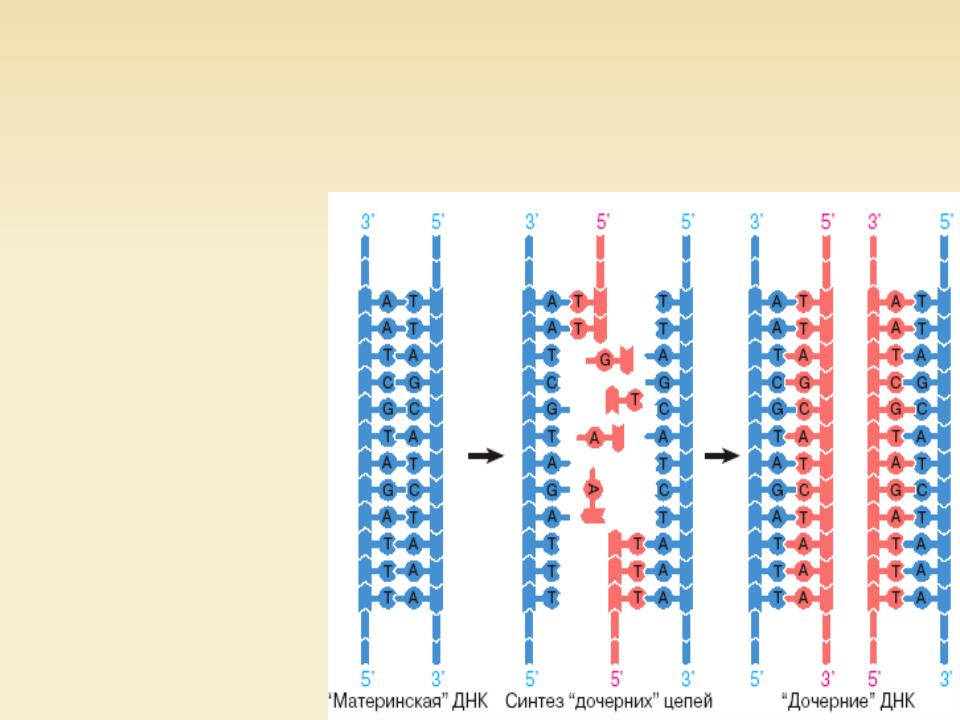 Вот почему репликация ДНК описывается как полуконсервативная, половина цепи является частью исходной молекулы ДНК, половина — совершенно новой.
Вот почему репликация ДНК описывается как полуконсервативная, половина цепи является частью исходной молекулы ДНК, половина — совершенно новой.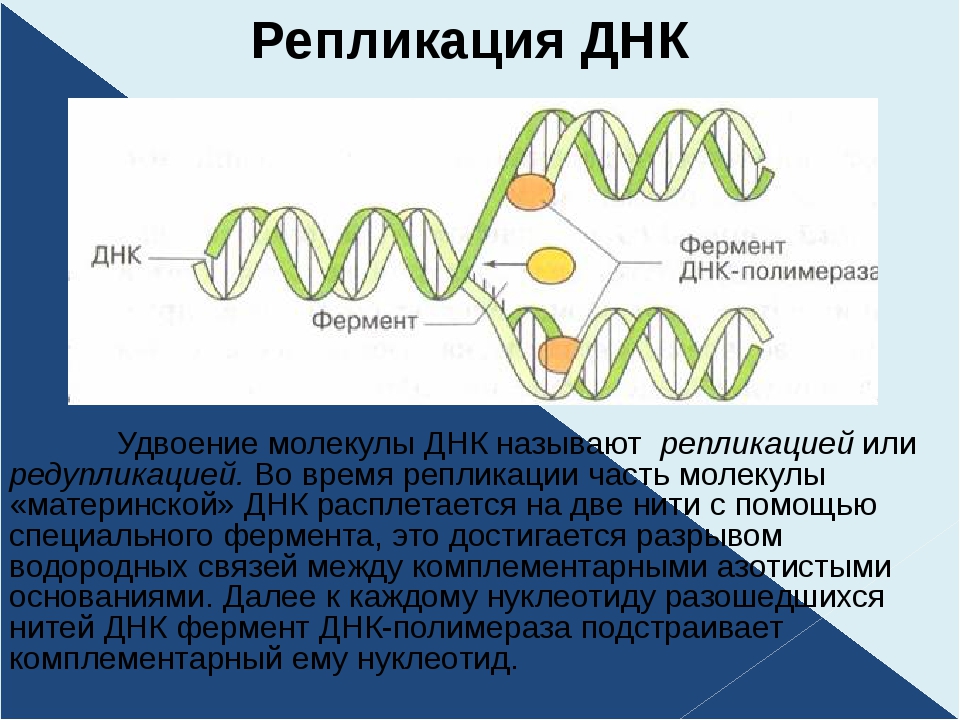 Он образует репликационную вилку, разрывая водородные связи между парами нуклеотидов в ДНК.
Он образует репликационную вилку, разрывая водородные связи между парами нуклеотидов в ДНК.